通过LLM掌握客户细分
简介
客户细分项目可以采用多种方法。在本文中,我将教你高级技巧,不仅可以定义聚类,还可以分析结果。
“在这篇文章中,我们会看到什么?”
我们将在本文中看到以下3种处理这类项目的方法:
1. Kmeans算法
2. K-Prototype算法
3. LLM + Kmeans算法
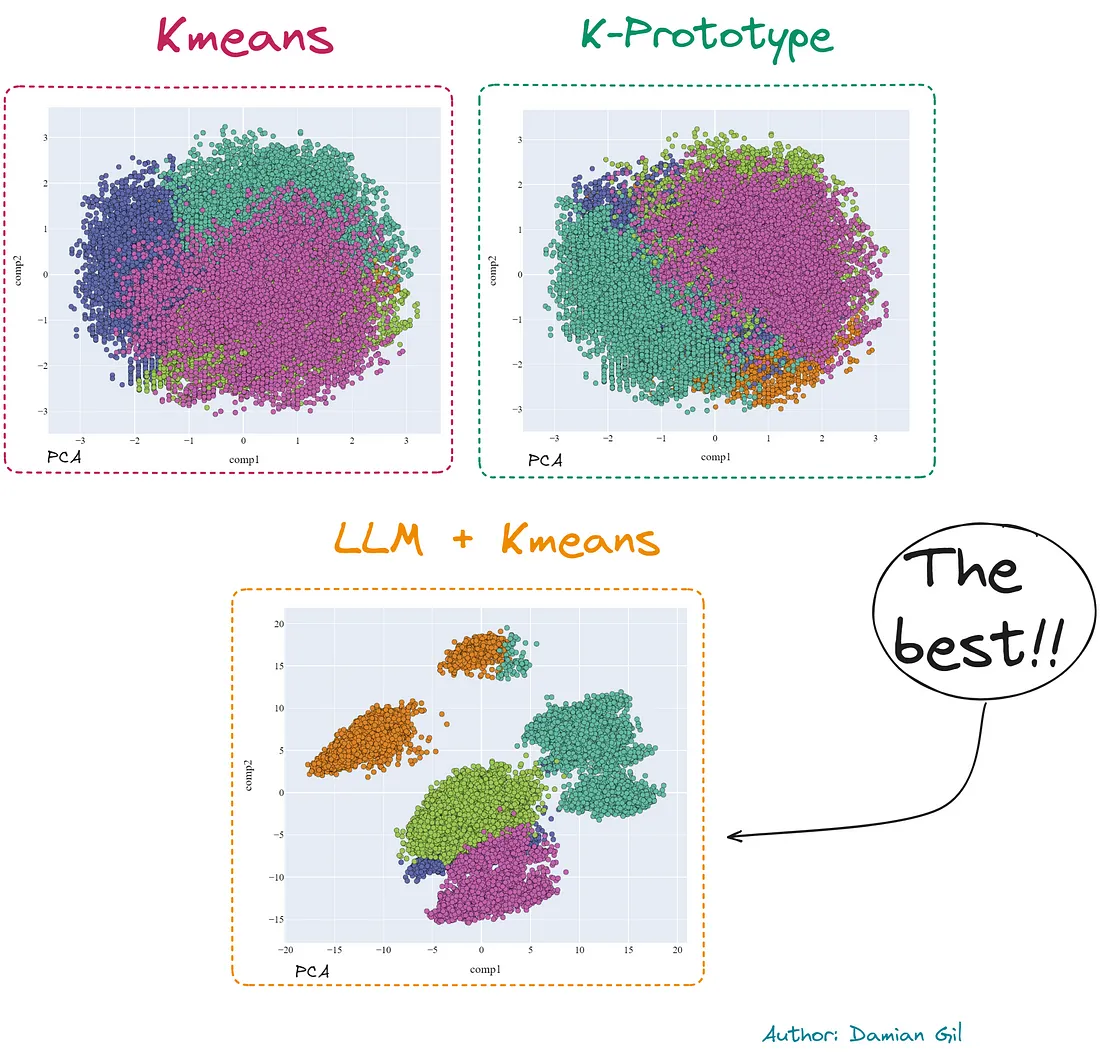
作为一个小预览,我将展示不同模型创建的2D表示(PCA)的比较。
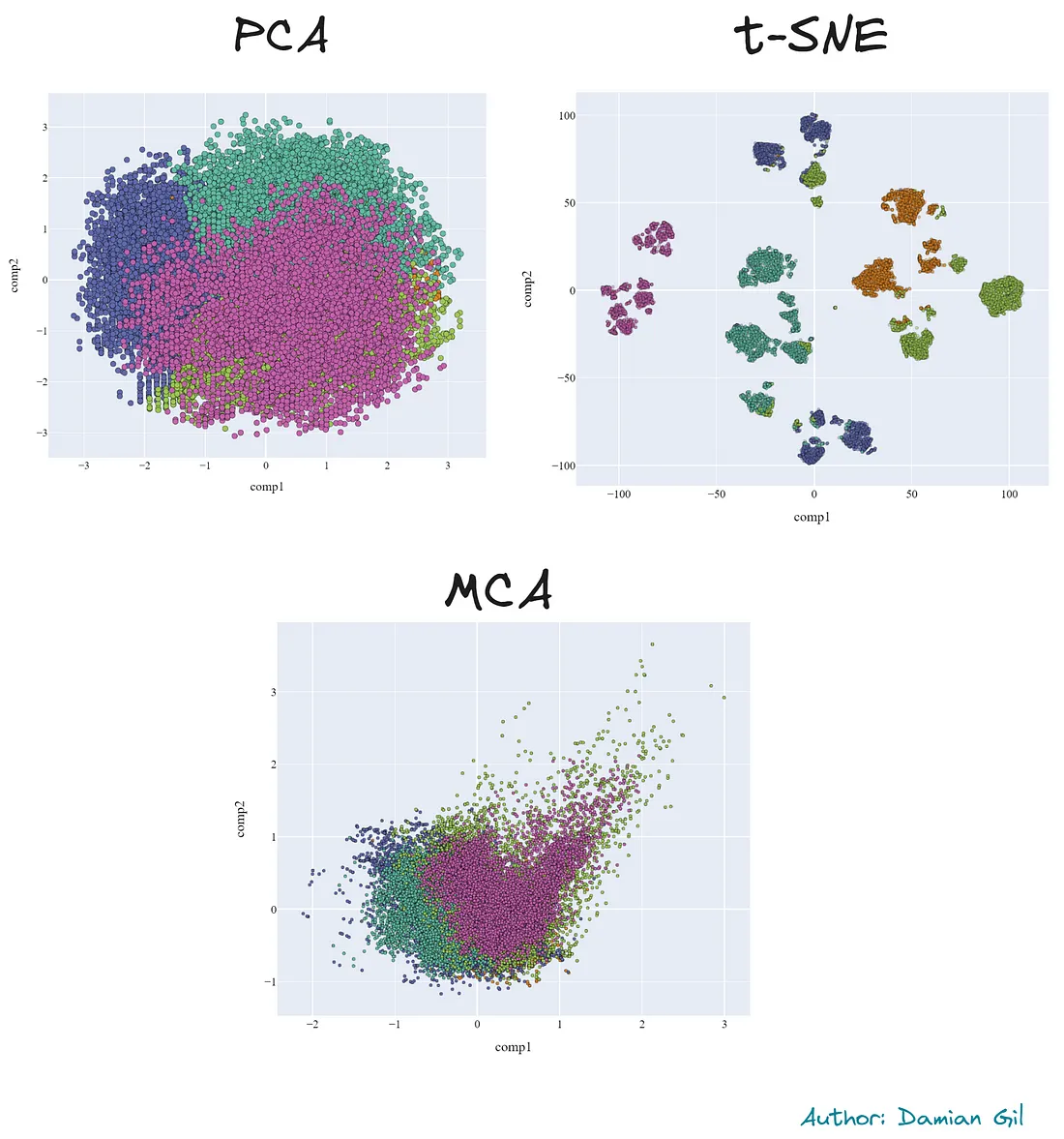
你还将学习降维技术,如:
1. PCA(主成分分析)
2. t-SNE(t-分布随机邻域嵌入)
3. MCA(多重对应分析)
其中部分结果如下:
数据
这个项目使用的原始数据来自公共Kaggle:银行数据集-营销目标。该数据集中的每一行都包含有关公司客户的信息。一些字段是数值型的,而其他字段是分类的,我们将看到这扩展了解决问题的可能方式。
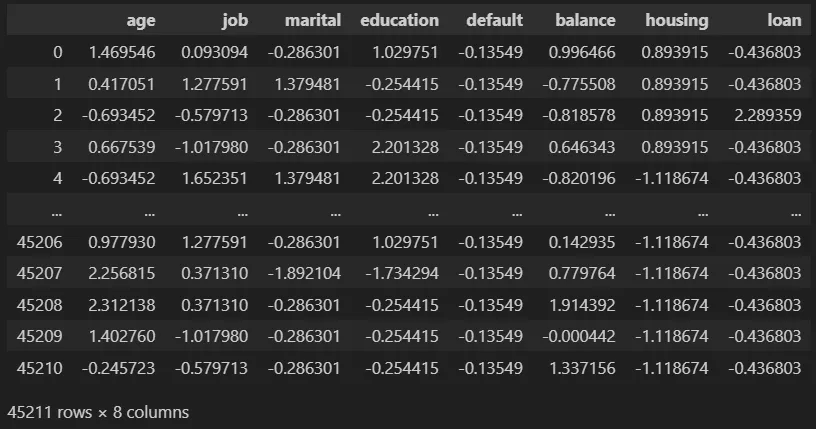
我们只保留前8行。我们的数据集是这样的:
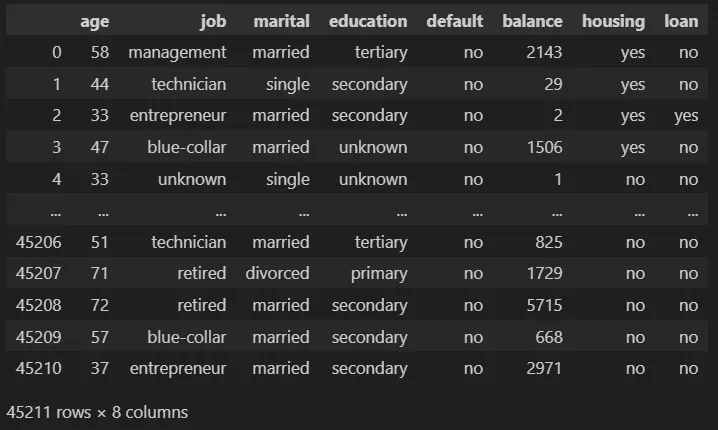
让我们看一下数据集中各列的简要描述:
1. age(数值型)
2. job:工作类型(分类变量:“管理人员”、“未知”、“失业”、“管理人员”、“家政婦”、“企业家”、“学生”、“蓝领”、“自雇”、“退休”、“技术员”、“服务”)
3. marital:婚姻状态(分类变量:“已婚”、“离婚”、“单身”;注意:“离婚”表示离异或丧偶)
4. education:(分类变量:“未知”、“中学”、“小学”、“大学”)
5. default:是否违约信用?(二进制变量:“是”、“否”)
6. balance:平均年度余额,以欧元为单位(数值型)
7. housing:是否有住房贷款?(二进制变量:“是”、“否”)
8. loan:是否有个人贷款?(二进制变量:“是”、“否”)
对于这个项目,我使用了Kaggle提供的训练数据集。可以找到一个名为“data”的文件夹,其中存储了一个压缩文件,其中包含项目中使用的数据集。另外,你将在压缩文件内找到两个CSV文件。一个是Kaggle提供的训练数据集(train.csv),另一个是执行嵌入后的数据集(embedding_train.csv),我们稍后会详细解释。
为了进一步澄清项目的结构,项目树如下所示:
clustering_llm
├─ datadata
│ ├─ data.rar
├─ img
├─ embedding.ipynb
├─ embedding_creation.py
├─ kmeans.ipynb
├─ kprototypes.ipynb
├─ README.md
└─ requirements.txt
方法1:Kmeans
这是最常见的方法,你肯定会知道。我们将研究它,因为在这些情况下,我将展示高级分析技术。
预处理
对变量进行预处理:
1. 将分类变量转换为数值变量。我们可以应用Onehot 编码器(通常做法),但在这种情况下,我们将应用Ordinal编码器。
2. 我们尝试确保数值变量具有高斯分布。为此,我们将应用PowerTransformer。
让我们看看代码中的效果。
import pandas as pd # dataframe manipulation
import numpy as np # linear algebra
# data visualization
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import plotly.express as px
import plotly.graph_objects as go
import seaborn as sns
import shap
# sklearn
from sklearn.cluster import KMeans
from sklearn.preprocessing import PowerTransformer, OrdinalEncoder
from sklearn.pipeline import Pipeline
from sklearn.manifold import TSNE
from sklearn.metrics import silhouette_score, silhouette_samples, accuracy_score, classification_report
from pyod.models.ecod import ECOD
from yellowbrick.cluster import KElbowVisualizer
import lightgbm as lgb
import prince
df = pd.read_csv("train.csv", sep = ";")
df = df.iloc[:, 0:8]
pipe = Pipeline([('ordinal', OrdinalEncoder()), ('scaler', PowerTransformer())])
pipe_fit = pipe.fit(df)
data = pd.DataFrame(pipe_fit.transform(df), columns = df.columns)
data
输出:
异常值
对于Kmeans算法而言,数据中的异常值越少越关键。我们可以应用常规的使用z得分来选择离群值的方法,但在本文中,我将向你展示一种更先进和酷的方法。
好的,那么这个方法是什么呢?我们将使用Python的异常值检测(PyOD)库。该库专注于检测不同案例的离群值。具体来说,我们将使用ECOD方法("基于经验累积分布函数的离群值检测")。
该方法旨在获取数据的分布,并了解概率密度较低(异常值)的值是哪些。
from pyod.models.ecod import ECOD
clf = ECOD()
clf.fit(data)
outliers = clf.predict(data)
data["outliers"] = outliers"outliers"] = outliers
# Data without outliers
data_no_outliers = data[data["outliers"] == 0]
data_no_outliers = data_no_outliers.drop(["outliers"], axis = 1)
# Data with Outliers
data_with_outliers = data.copy()
data_with_outliers = data_with_outliers.drop(["outliers"], axis = 1)
print(data_no_outliers.shape) -> (40691, 8)
print(data_with_outliers.shape) -> (45211, 8)
建模
使用Kmeans算法的一个缺点是必须选择要使用的聚类数。在这种情况下,为了获得该数据,我们将使用Elbow Method。它包括计算簇内点与其质心之间的畸变程度。目标很明确,即获得尽可能小的畸变。在这种情况下,我们使用以下代码:
from yellowbrick.cluster import KElbowVisualizer
# Instantiate the clustering model and visualizer
km = KMeans(init="k-means++", random_state=0, n_init="auto")
visualizer = KElbowVisualizer(km, k=(2,10))
visualizer.fit(data_no_outliers) # Fit the data to the visualizer
visualizer.show()
输出:
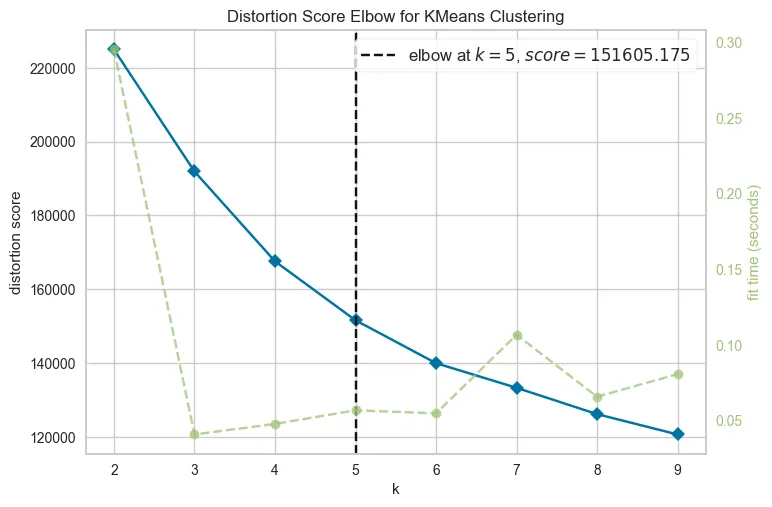
我们可以从k=5开始看到,失真度变化不会出现剧烈的变动。理想情况下,从k=5开始的行为应该几乎是稳定的。但这种情况很少发生,所以可以采用其他方法来确定最优的聚类数量。为了确认,我们可以进行Silhoutte可视化。代码如下:
from sklearn.metrics import davies_bouldin_score, silhouette_score, silhouette_samples
import matplotlib.cm as cm
def make_Silhouette_plot(X, n_clusters):
plt.xlim([-0.1, 1])
plt.ylim([0, len(X) + (n_clusters + 1) * 10])
clusterer = KMeans(n_clusters=n_clusters, max_iter = 1000, n_init = 10, init = 'k-means++', random_state=10)
cluster_labels = clusterer.fit_predict(X)
silhouette_avg = silhouette_score(X, cluster_labels)
print(
"For n_clusters =", n_clusters,
"The average silhouette_score is :", silhouette_avg,
)
# Compute the silhouette scores for each sample
sample_silhouette_values = silhouette_samples(X, cluster_labels)
y_lower = 10
for i in range(n_clusters):
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_clusters)
plt.fill_betweenx(
np.arange(y_lower, y_upper),
0,
ith_cluster_silhouette_values,
facecolor=color,
edgecolor=color,
alpha=0.7,
)
plt.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10
plt.title(f"The Silhouette Plot for n_cluster = {n_clusters}", fontsize=26)
plt.xlabel("The silhouette coefficient values", fontsize=24)
plt.ylabel("Cluster label", fontsize=24)
plt.axvline(x=silhouette_avg, color="red", linestyle="--")
plt.yticks([])
plt.xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
range_n_clusters = list(range(2,10))
for n_clusters in range_n_clusters:
print(f"N cluster: {n_clusters}")
make_Silhouette_plot(data_no_outliers, n_clusters)
plt.savefig('Silhouette_plot_{}.png'.format(n_clusters))
plt.close()
OUTPUT:
"""
N cluster: 2
For n_clusters = 2 The average silhouette_score is : 0.1775761520337095
N cluster: 3
For n_clusters = 3 The average silhouette_score is : 0.20772622268785523
N cluster: 4
For n_clusters = 4 The average silhouette_score is : 0.2038116470937145
N cluster: 5
For n_clusters = 5 The average silhouette_score is : 0.20142888327171368
N cluster: 6
For n_clusters = 6 The average silhouette_score is : 0.20252892716996912
N cluster: 7
For n_clusters = 7 The average silhouette_score is : 0.21185490763840265
N cluster: 8
For n_clusters = 8 The average silhouette_score is : 0.20867816457291538
N cluster: 9
For n_clusters = 9 The average silhouette_score is : 0.21154289421300868
"""
可以看到,当n_cluster=9时,得到的轮廓系数最高,但与其他得分相比,得分变化很小。目前,前面的结果并没有为我们提供太多信息。另一方面,前面的代码创建了轮廓可视化,为我们提供了更多信息。
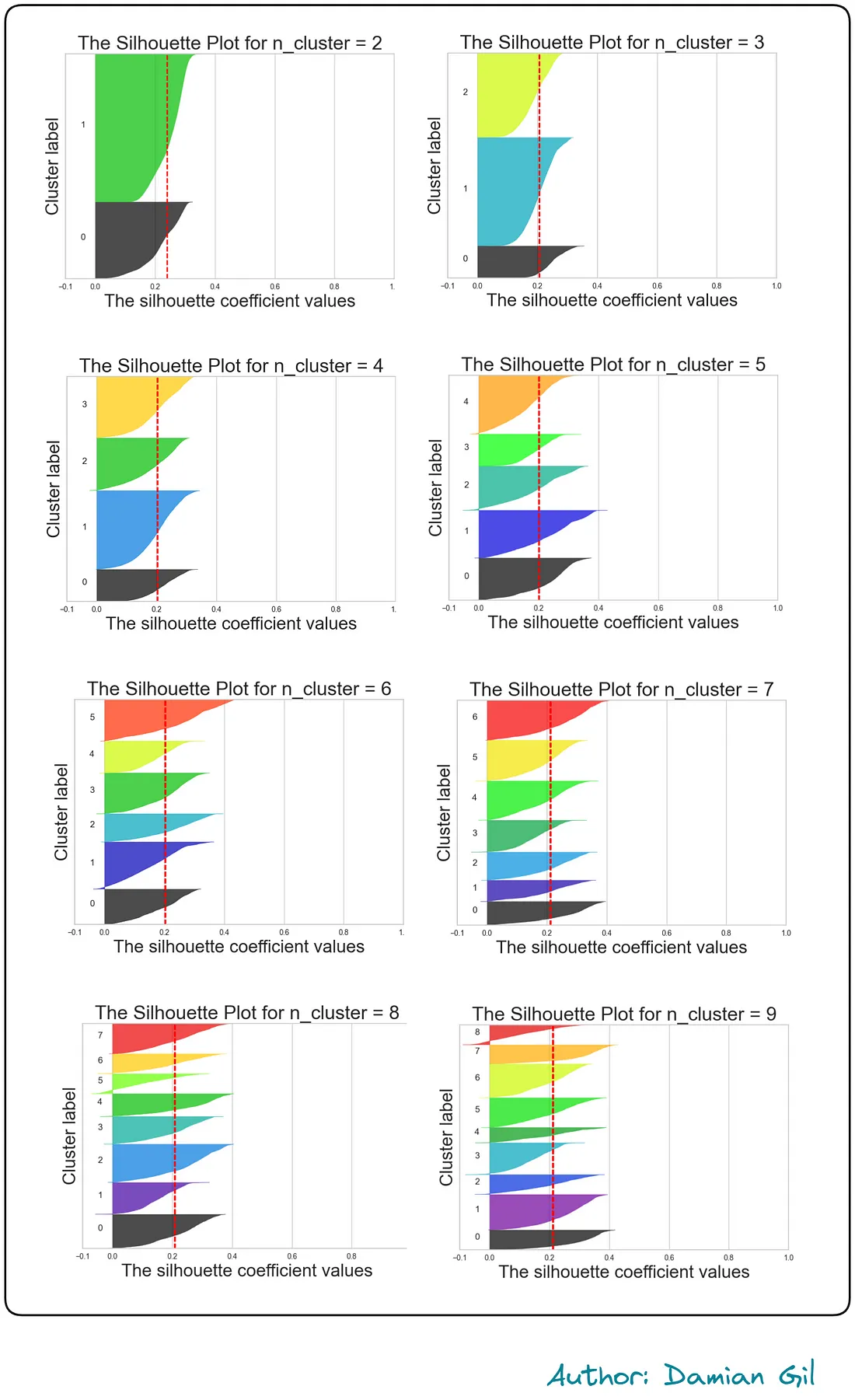
由于理解这些表示并不是本文的目标,我得出结论,似乎没有一个非常明确的决策来确定哪个数字是最好的。在查看之前的表示后,我们可以选择K=5或K=6。这是因为对于不同的簇,它们的轮廓系数得分高于平均值,并且簇的大小没有不平衡的情况。
最后,我们可以用K = 5创建我们的Kmeans模型。
km = KMeans(n_clusters=5,5,
init='k-means++',
n_init=10,
max_iter=100,
random_state=42)
clusters_predict = km.fit_predict(data_no_outliers)
"""
clusters_predict -> array([4, 2, 0, ..., 3, 4, 3])
np.unique(clusters_predict) -> array([0, 1, 2, 3, 4])
"""
评估
Kmeans模型的方法相对于其他模型更为开放。我们可以使用以下方式进行评估:
1. 指标(metrics)
2. 可视化(visualizations)
3. 解释(对于公司来说非常重要)
关于模型评估指标,我们可以使用以下代码:
from sklearn.metrics import silhouette_score
from sklearn.metrics import calinski_harabasz_score
from sklearn.metrics import davies_bouldin_score
"""
The Davies Bouldin index is defined as the average similarity measure
of each cluster with its most similar cluster, where similarity
is the ratio of within-cluster distances to between-cluster distances.
The minimum value of the DB Index is 0, whereas a smaller
value (closer to 0) represents a better model that produces better clusters.
"""
print(f"Davies bouldin score: {davies_bouldin_score(data_no_outliers,clusters_predict)}")
"""
Calinski Harabaz Index -> Variance Ratio Criterion.
Calinski Harabaz Index is defined as the ratio of the
sum of between-cluster dispersion and of within-cluster dispersion.
The higher the index the more separable the clusters.
"""
print(f"Calinski Score: {calinski_harabasz_score(data_no_outliers,clusters_predict)}")
"""
The silhouette score is a metric used to calculate the goodness of
fit of a clustering algorithm, but can also be used as
a method for determining an optimal value of k (see here for more).
Its value ranges from -1 to 1.
A value of 0 indicates clusters are overlapping and either
the data or the value of k is incorrect.
1 is the ideal value and indicates that clusters are very
dense and nicely separated.
"""
print(f"Silhouette Score: {silhouette_score(data_no_outliers,clusters_predict)}")
OUTPUT:
"""
Davies bouldin score: 1.5480952939773156
Calinski Score: 7646.959165727562
Silhouette Score: 0.2013600389183821
"""
据所展示的情况,我们没有一个过度出色的模型。Davies的评分告诉我们,聚类之间的距离相当小。
这可能由于几个因素,但要记住,模型的能量就是数据;如果数据没有足够的预测能力,你就不能期望取得异常的结果。
对于可视化,我们可以使用降维方法PCA。为此,我们将使用专注于探索性分析和降维的Prince库。
首先,我们将计算三维主要分量,然后进行表示。这是前面步骤中执行的两个函数。
import prince
import plotly.express as px
def get_pca_2d(df, predict):
pca_2d_object = prince.PCA(
n_components=2,
n_iter=3,
rescale_with_mean=True,
rescale_with_std=True,
copy=True,
check_input=True,
engine='sklearn',
random_state=42
)
pca_2d_object.fit(df)
df_pca_2d = pca_2d_object.transform(df)
df_pca_2d.columns = ["comp1", "comp2"]
df_pca_2d["cluster"] = predict
return pca_2d_object, df_pca_2d
def get_pca_3d(df, predict):
pca_3d_object = prince.PCA(
n_components=3,
n_iter=3,
rescale_with_mean=True,
rescale_with_std=True,
copy=True,
check_input=True,
engine='sklearn',
random_state=42
)
pca_3d_object.fit(df)
df_pca_3d = pca_3d_object.transform(df)
df_pca_3d.columns = ["comp1", "comp2", "comp3"]
df_pca_3d["cluster"] = predict
return pca_3d_object, df_pca_3d
def plot_pca_3d(df, title = "PCA Space", opacity=0.8, width_line = 0.1):
df = df.astype({"cluster": "object"})
df = df.sort_values("cluster")
fig = px.scatter_3d(
df,
x='comp1',
y='comp2',
z='comp3',
color='cluster',
template="plotly",
# symbol = "cluster",
color_discrete_sequence=px.colors.qualitative.Vivid,
title=title).update_traces(
# mode = 'markers',
marker={
"size": 4,
"opacity": opacity,
# "symbol" : "diamond",
"line": {
"width": width_line,
"color": "black",
}
}
).update_layout(
width = 800,
height = 800,
autosize = True,
showlegend = True,
legend=dict(title_font_family="Times New Roman",
font=dict(size= 20)),
scene = dict(xaxis=dict(title = 'comp1', titlefont_color = 'black'),
yaxis=dict(title = 'comp2', titlefont_color = 'black'),
zaxis=dict(title = 'comp3', titlefont_color = 'black')),
font = dict(family = "Gilroy", color = 'black', size = 15))
fig.show()
不要太担心那些功能,按照以下方式使用它们:
pca_3d_object, df_pca_3d = pca_plot_3d(data_no_outliers, clusters_predict)
plot_pca_3d(df_pca_3d, title = "PCA Space", opacity=1, width_line = 0.1)"PCA Space", opacity=1, width_line = 0.1)
print("The variability is :", pca_3d_object.eigenvalues_summary)
输出:
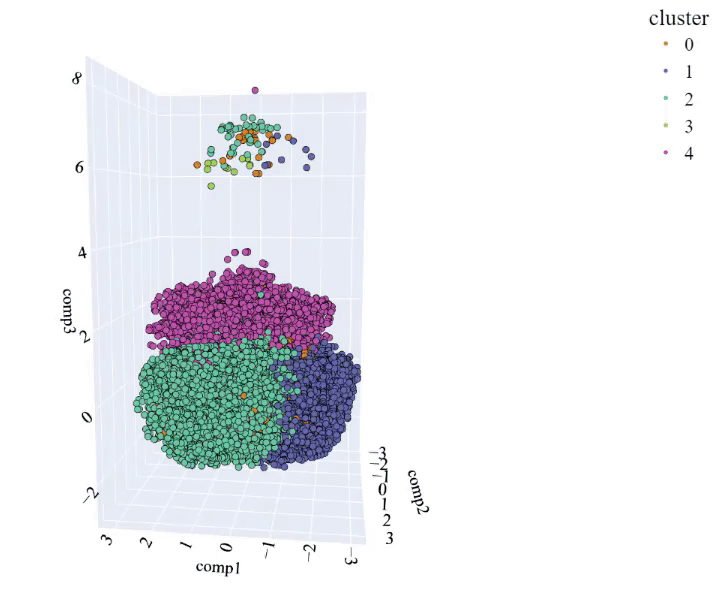
可以看到,这些聚类之间几乎没有分离,并且没有明显的界限,这与指标提供的信息是一致的。
需要注意的是,很少有人注意到主成分分析(PCA)和特征向量的变异性。
假设每个特征包含一定量的信息,它们各自为聚类提供信息。如果三个主要成分的累积和达到了80%的变异性,我们可以说结果是可接受的,表示法得到了良好的结果。如果这个值更低,我们必须将可视化结果持保留态度,因为我们丢失了其他特征向量中包含的许多信息。
下一个问题很明显:执行PCA的变异性是多少?
答案如下:
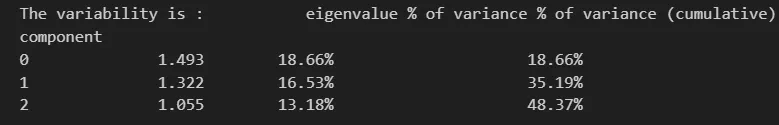
正如可以看到的那样,我们在前三个成分中有48.37%的可变性,这不足以得出明确结论。
事实证明,在运行PCA分析时,并没有保留空间结构。幸运的是,还有一个不太知名的方法叫做t-SNE,可以减少维度并且保持空间结构。这可以帮助我们进行可视化,因为之前的方法并没有取得太大成功。
如果你在计算机上尝试运行它,请记住它有更高的计算成本。因此,我对原始数据集进行了抽样,仍然需要大约5分钟才能得到结果。代码如下:
from sklearn.manifold import TSNE
sampling_data = data_no_outliers.sample(frac=0.5, replace=True, random_state=1)
sampling_clusters = pd.DataFrame(clusters_predict).sample(frac=0.5, replace=True, random_state=1)[0].values
df_tsne_3d = TSNE(
n_components=3,
learning_rate=500,
init='random',
perplexity=200,
n_iter = 5000).fit_transform(sampling_data)
df_tsne_3d = pd.DataFrame(df_tsne_3d, columns=["comp1", "comp2",'comp3'])
df_tsne_3d["cluster"] = sampling_clusters
plot_pca_3d(df_tsne_3d, title = "PCA Space", opacity=1, width_line = 0.1)
作为结果,我得到了下面的图像。它显示了簇之间更大的分离,并使我们能够以更清晰的方式得出结论。
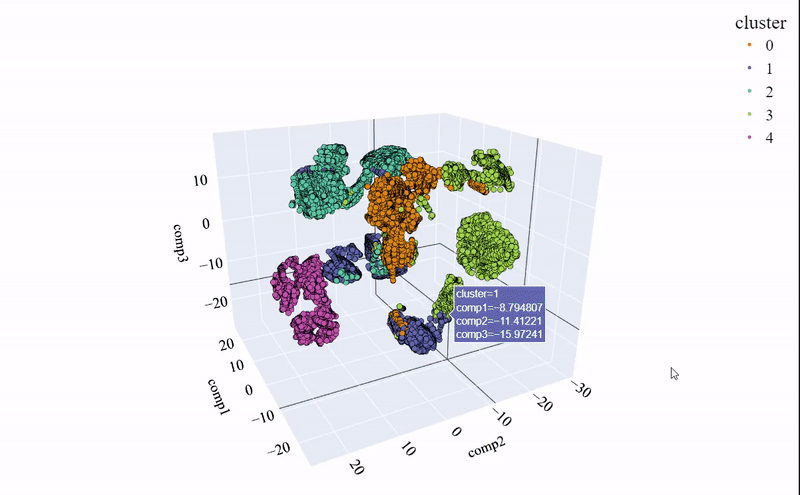
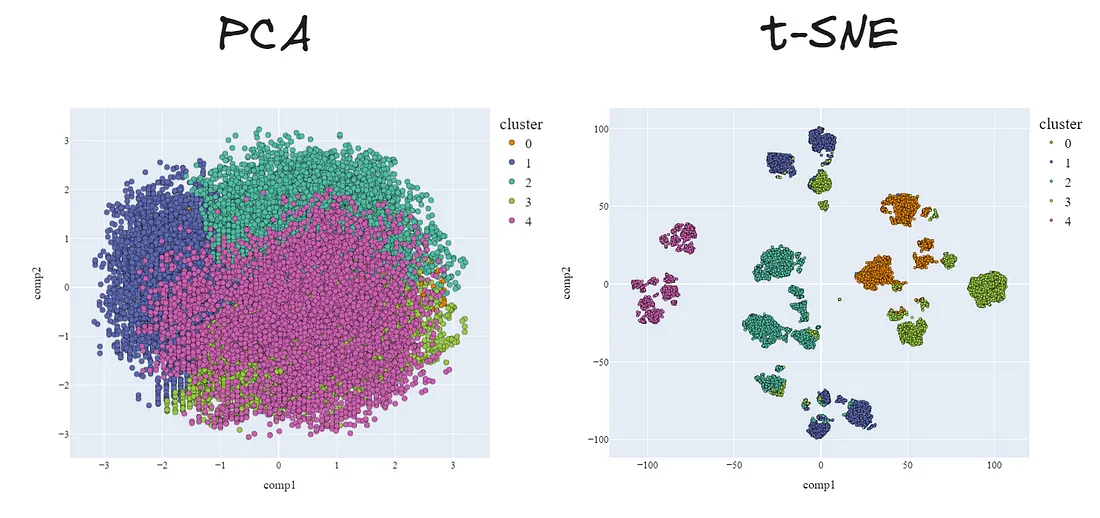
实际上,我们可以比较主成分分析(PCA)和t-SNE在2维下进行的降维。使用第二种方法可以明显改进。
最后,让我们稍微探讨一下模型的工作原理,了解哪些特征最重要以及簇的主要特征。
为了看到每个变量的重要性,在这种情况下,我们将使用一种常见的“技巧”。我们将创建一个分类模型,其中“X”是Kmeans模型的输入,“y”是Kmeans模型预测的簇。
选择的模型是LGBMClassifier。这个模型非常强大,在处理分类和数值变量时效果好。通过使用SHAP库训练这个新模型,我们可以获得每个特征在预测中的重要性。代码如下:
import lightgbm as lgb
import shap
# We create the LGBMClassifier model and train it
clf_km = lgb.LGBMClassifier(colsample_by_tree=0.8)
clf_km.fit(X=data_no_outliers, y=clusters_predict)
#SHAP values
explainer_km = shap.TreeExplainer(clf_km)
shap_values_km = explainer_km.shap_values(data_no_outliers)
shap.summary_plot(shap_values_km, data_no_outliers, plot_type="bar", plot_size=(15, 10))
输出:
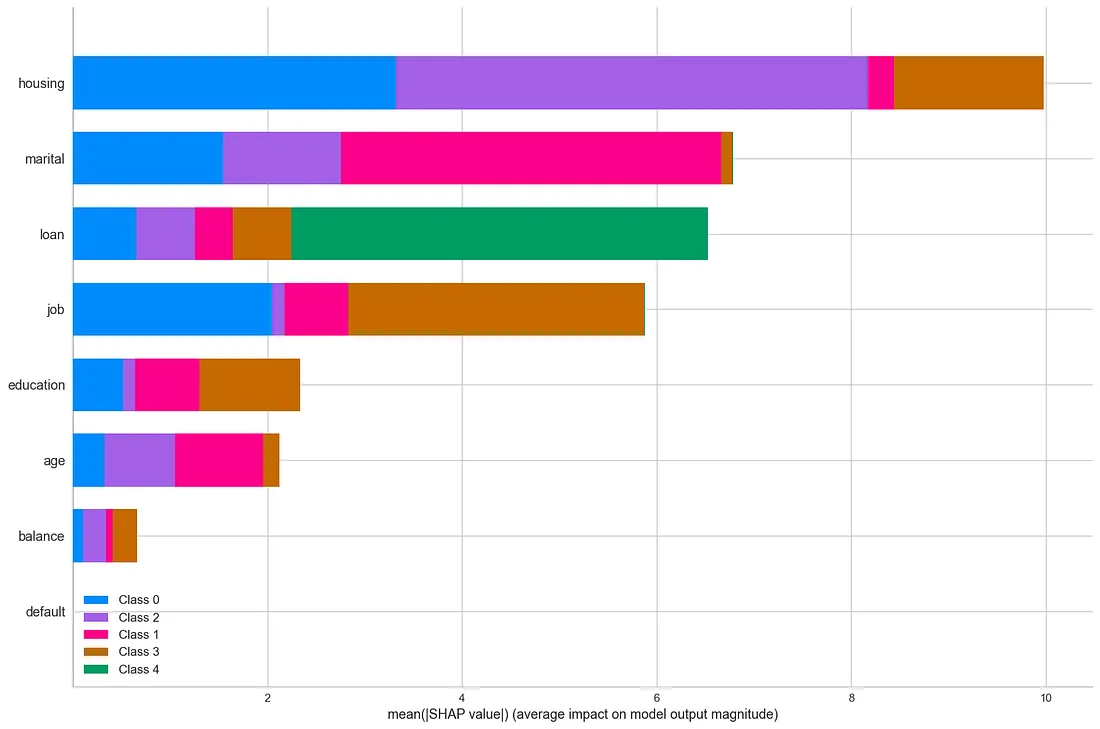
可以看出,特征房屋拥有最强的预测能力。同时也可以看出,集群编号4(绿色)主要通过贷款变量进行区分。
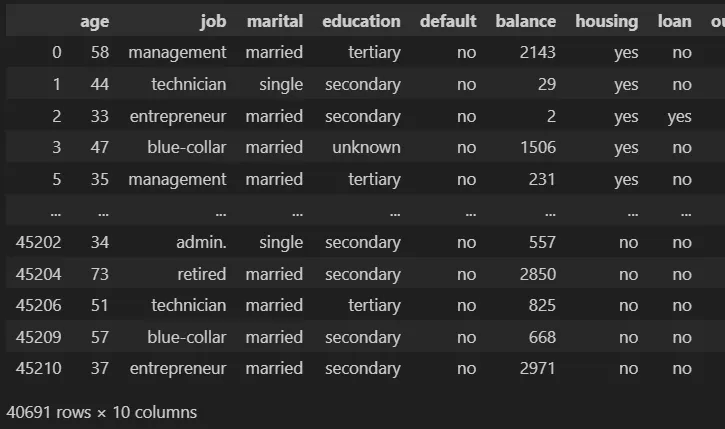
最后,我们必须对集群的特征进行分析。这部分研究对于业务至关重要。我们将为每个集群的数据集特征获取均值(对于数值变量)和最频繁的值(对于分类变量)。
df_no_outliers = df[df.outliers == 0]0]
df_no_outliers["cluster"] = clusters_predict
df_no_outliers.groupby('cluster').agg(
{
'job': lambda x: x.value_counts().index[0],
'marital': lambda x: x.value_counts().index[0],
'education': lambda x: x.value_counts().index[0],
'housing': lambda x: x.value_counts().index[0],
'loan': lambda x: x.value_counts().index[0],
'contact': lambda x: x.value_counts().index[0],
'age':'mean',
'balance': 'mean',
'default': lambda x: x.value_counts().index[0],
}
).reset_index()
输出:
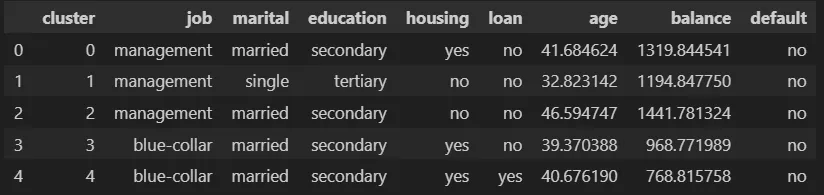
我们看到,职业为蓝领工人的聚类之间在特征上没有很大的差异。这是不希望出现的情况,因为难以区分每个聚类的客户。而当职业为管理人员时,我们可以获得更好的区分。
在以不同方式进行分析后,它们得出了相同的结论:“我们需要改进结果”。
方法2:K-Prototype
如果我们记得我们的原始数据集,我们可以看到我们有分类和数值变量。不幸的是,由Skelearn提供的Kmeans算法不接受分类变量,迫使原始数据集进行修改和重大改变。
预处理
由于我们有数值变量,我们必须对它们进行某些修改。通常建议所有数值变量都在类似的尺度上,并且分布尽可能接近高斯分布。我们将用于创建模型的数据集如下创建:
pipe = Pipeline([('scaler', PowerTransformer())])'scaler', PowerTransformer())])
df_aux = pd.DataFrame(pipe_fit.fit_transform(df_no_outliers[["age", "balance"]] ), columns = ["age", "balance"])
df_no_outliers_norm = df_no_outliers.copy()
# Replace age and balance columns by preprocessed values
df_no_outliers_norm = df_no_outliers_norm.drop(["age", "balance"], axis = 1)
df_no_outliers_norm["age"] = df_aux["age"].values
df_no_outliers_norm["balance"] = df_aux["balance"].values
df_no_outliers_norm
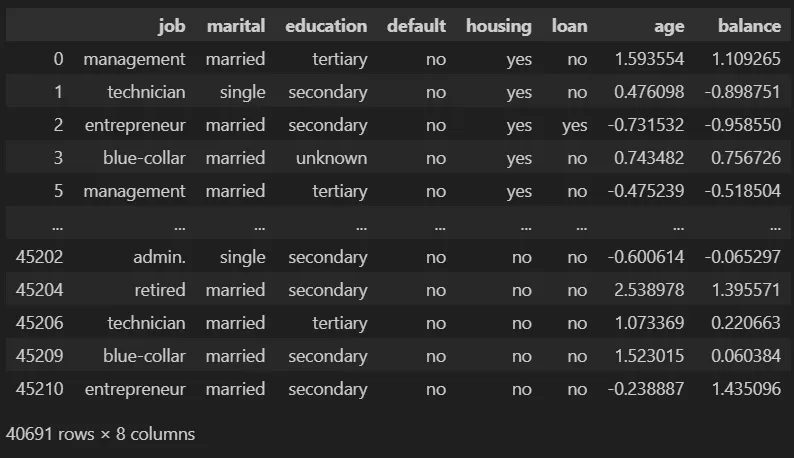
异常值
由于我所提出的异常值检测方法(ECOD)只接受数值变量,因此必须进行与k均值方法相同的转换。我们应用异常值检测模型,该模型将告诉我们哪些行需要被消除,最终得到我们将用作K-Prototype模型输入的数据集。
建模
我们创建模型,为此首先需要得到最佳的k值。为了做到这一点,我们使用“拐点法”和以下代码。
# Choose optimal K using Elbow method
from kmodes.kprototypes import KPrototypes
from plotnine import *
import plotnine
cost = []
range_ = range(2, 15)
for cluster in range_:
kprototype = KPrototypes(n_jobs = -1, n_clusters = cluster, init = 'Huang', random_state = 0)
kprototype.fit_predict(df_no_outliers, categorical = categorical_columns_index)
cost.append(kprototype.cost_)
print('Cluster initiation: {}'.format(cluster))
# Converting the results into a dataframe and plotting them
df_cost = pd.DataFrame({'Cluster':range_, 'Cost':cost})
# Data viz
plotnine.options.figure_size = (8, 4.8)
(
ggplot(data = df_cost)+
geom_line(aes(x = 'Cluster',
y = 'Cost'))+
geom_point(aes(x = 'Cluster',
y = 'Cost'))+
geom_label(aes(x = 'Cluster',
y = 'Cost',
label = 'Cluster'),
size = 10,
nudge_y = 1000) +
labs(title = 'Optimal number of cluster with Elbow Method')+
xlab('Number of Clusters k')+
ylab('Cost')+
theme_minimal()
)
输出:
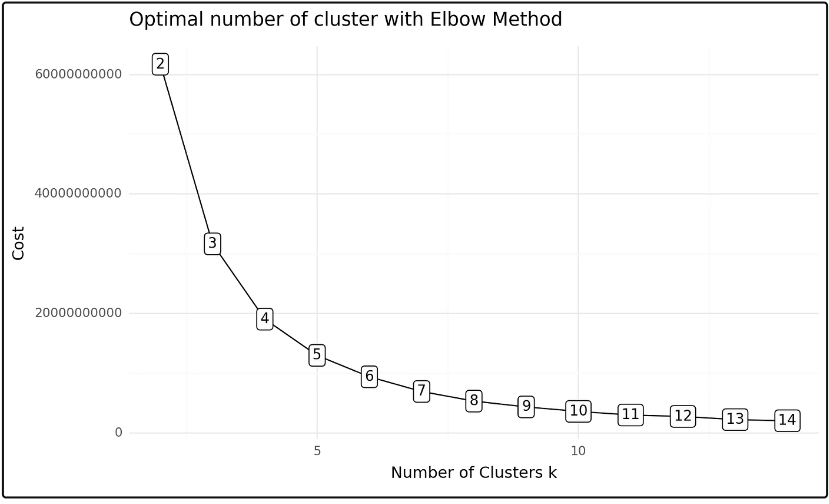
我们可以看到最佳选择是K=5。
请注意,由于该算法所需时间比通常使用的算法稍长,对于前一个图,需要耗时86分钟,请记住这一点。

好的,我们现在已经明确了聚类的数量,我们只需要创建模型。
# We get the index of categorical columns
numerics = ['int16', 'int32', 'int64', 'float16', 'float32', 'float64']
categorical_columns = df_no_outliers_norm.select_dtypes(exclude=numerics).columns
print(categorical_columns)
categorical_columns_index = [df_no_outliers_norm.columns.get_loc(col) for col in categorical_columns]
# Create the model
cluster_num = 5
kprototype = KPrototypes(n_jobs = -1, n_clusters = cluster_num, init = 'Huang', random_state = 0)
kprototype.fit(df_no_outliers_norm, categorical = categorical_columns_index)
clusters = kprototype.predict(df_no_outliers , categorical = categorical_columns_index)
print(clusters) " -> array([3, 1, 1, ..., 1, 1, 2], dtype=uint16)"
我们已经拥有我们的模型和它的预测结果,我们只需要进行评估。
评估
正如我们之前所见,我们可以应用几种可视化方法来直观地了解我们的模型的好坏。不幸的是,PCA方法和t-SNE不接受分类变量。但是不用担心,因为Prince库包含MCA(多重对应分析)方法,并且它接受混合数据集。如下图所示:
计划是应用一个多元对应分析(MCA)来减少维度并能够制作图形表示。为此我们使用以下代码:
from prince import MCA
def get_MCA_3d(df, predict):
mca = MCA(n_components =3, n_iter = 100, random_state = 101)
mca_3d_df = mca.fit_transform(df)
mca_3d_df.columns = ["comp1", "comp2", "comp3"]
mca_3d_df["cluster"] = predict
return mca, mca_3d_df
def get_MCA_2d(df, predict):
mca = MCA(n_components =2, n_iter = 100, random_state = 101)
mca_2d_df = mca.fit_transform(df)
mca_2d_df.columns = ["comp1", "comp2"]
mca_2d_df["cluster"] = predict
return mca, mca_2d_df
"-------------------------------------------------------------------"
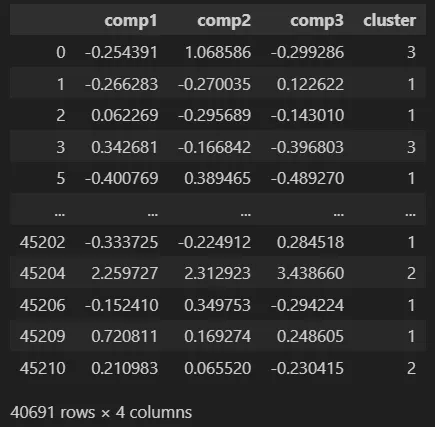
mca_3d, mca_3d_df = get_MCA_3d(df_no_outliers_norm, clusters)
数据集mca_3d_df包含了这些信息:
让我们使用多元对比分析(MCA)方法提供的降维数据来制定一个情节。
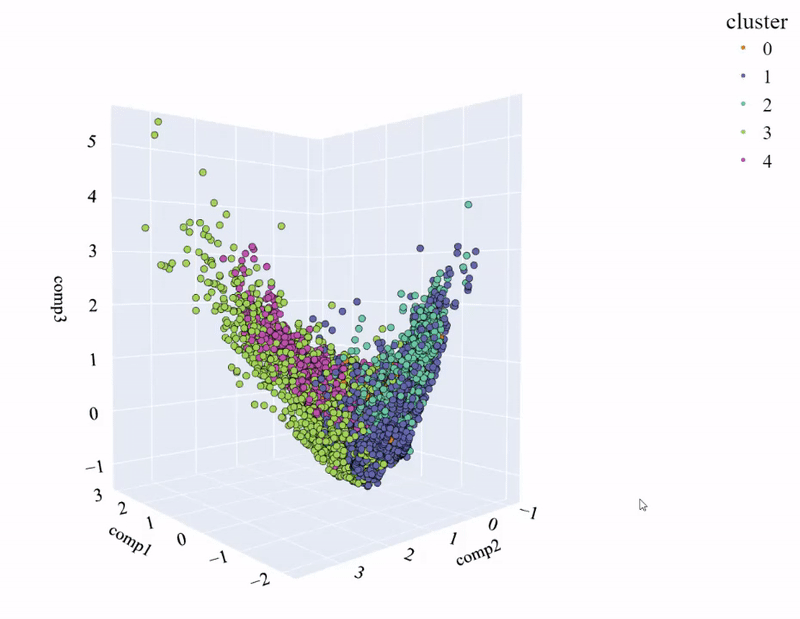
无法将聚类区分开来。那么我们可以说这个模型还不够好,对吗?
我希望你说了类似这样的话:
“嘿,达米安,别这么快就下结论啊!你有没有看到MCA提供的这三个组分的可变性呢?”
事实上,我们必须看一下前三个组分的变异性是否足够用来得出结论。MCA方法可以让我们以一种非常简单的方式得到这些值。
mca_3d.eigenvalues_summaryeigenvalues_summary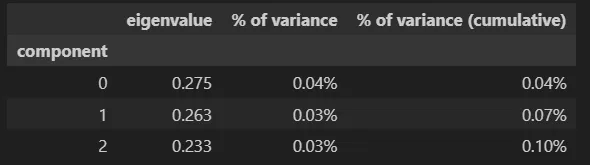
根据我们的数据,我们基本上得到了零的变异性。
换句话说,通过MCA提供的降维信息,我们无法得出清晰的结论来支持我们的模型。
通过展示这些结果,我试图举一个真实数据项目中发生的情况的例子。并不总能得到好的结果,但一个好的数据科学家知道如何识别原因。
我们有最后一个选项来通过可视化来判断K-Prototype方法创建的模型是否合适。这个过程很简单:
1. 将预处理后的数据集应用PCA,将分类变量转换为数值变量。
2. 获得PCA的主成分
3. 使用主成分来做一个表示,例如使用坐标轴和点的颜色来预测K-Prototype模型。
请注意,由PCA提供的主成分与方法1的Kmeans相同,因为它们使用的是相同的数据帧。
让我们看看我们得到了什么...
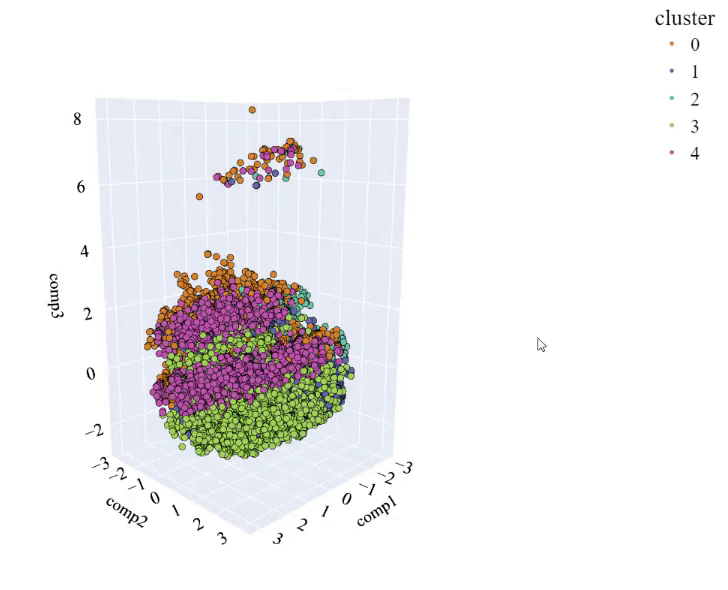
看起来并不糟糕,实际上它与Kmeans所得的结果有一定的相似性。
最后,我们获得了聚类的平均值和每个变量的重要性。
变量的权重最大的是数值变量,特别是考虑到这两个特征的限制几乎足以区分每个聚类。
简而言之,可以说得到了与Kmeans类似的结果。
方法3:LLM+Kmeans
LLM不能直接理解书面文本,我们需要将这类模型的输入转换为数值向量。为此,进行词嵌入。下面的图片可以解释这个概念。
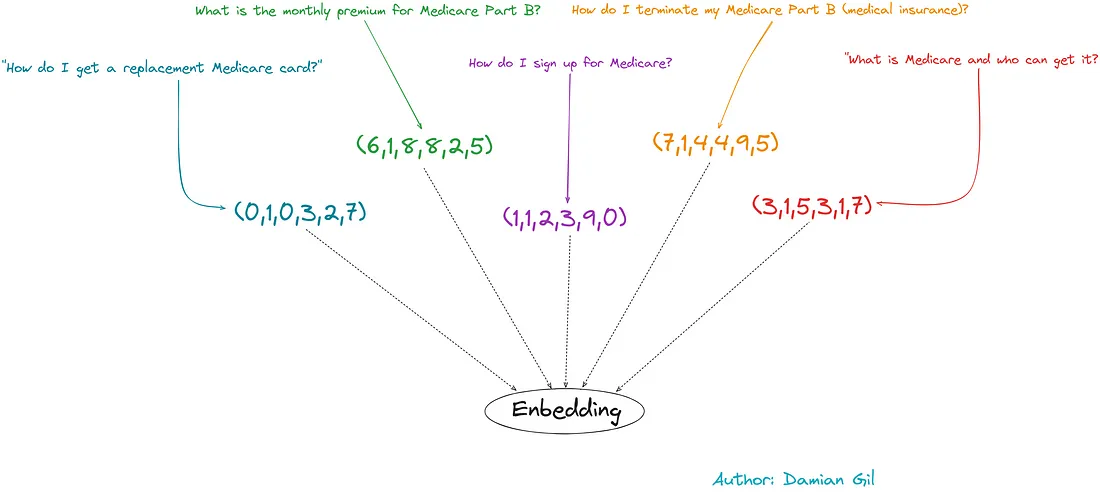
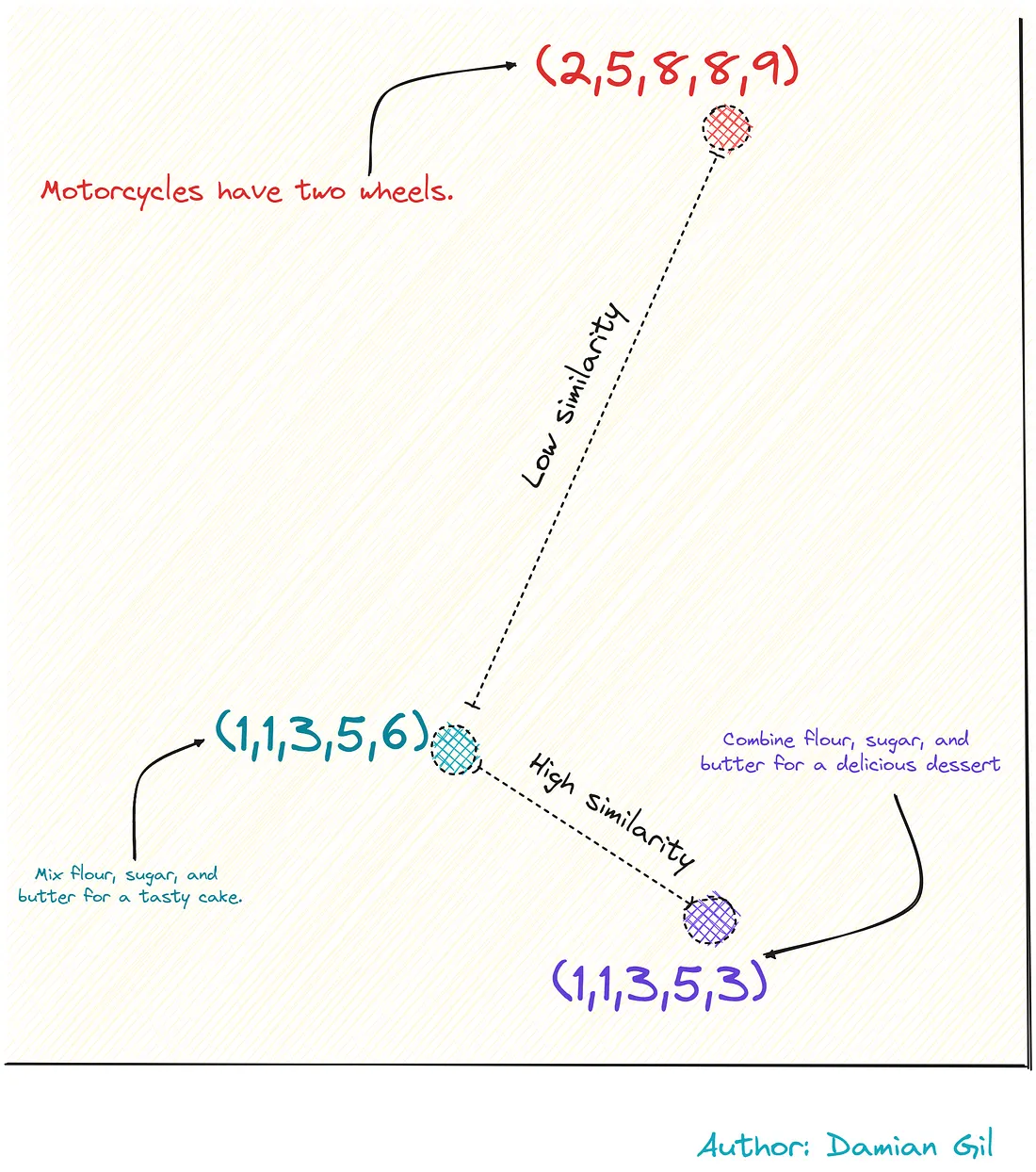
这种编码是智能完成的,也就是说,具有类似含义的短语将具有更相似的向量。请参考下面的图片。
Word embedding通过所谓的转换进行,这是一种专门用于编码的算法。通常你可以选择编码后的数字向量的大小。以下是其中一个关键点:
“由于嵌入得到的向量具有大量的维度,数据中的小变化可以更精确地被观察到。”
因此,如果我们将信息丰富的Kmeans模型输入,它将返回更好的预测结果。这就是我们追求的思路,以下是具体步骤:
1. 通过词嵌入来转换我们的原始数据集
2. 创建一个Kmeans模型
3. 评估模型
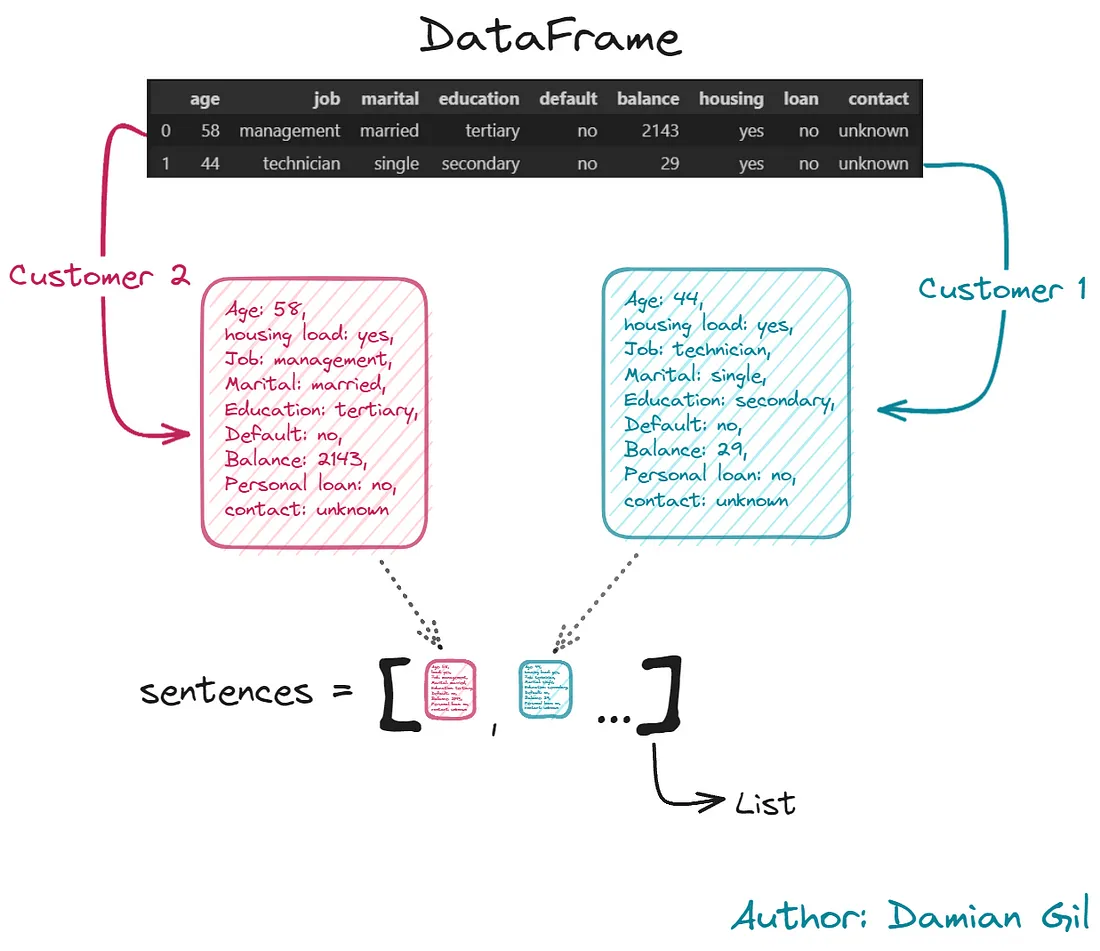
好的,第一步是通过词嵌入来编码信息。我们的目的是将每个客户的信息统一成包含其所有特征的文本。这部分需要很长的计算时间。所以我创建了一个名为embedding_creation.py的脚本来完成这项工作。该脚本收集训练数据集中的数值,并创建一个由嵌入提供的新数据集。以下是脚本代码:
import pandas as pd # dataframe manipulation
import numpy as np # linear algebra
from sentence_transformers import SentenceTransformer
df = pd.read_csv("data/train.csv", sep = ";")
# -------------------- First Step --------------------
def compile_text(x):
text = f"""Age: {x['age']},
housing load: {x['housing']},
Job: {x['job']},
Marital: {x['marital']},
Education: {x['education']},
Default: {x['default']},
Balance: {x['balance']},
Personal loan: {x['loan']},
contact: {x['contact']}
"""
return text
sentences = df.apply(lambda x: compile_text(x), axis=1).tolist()
# -------------------- Second Step --------------------
model = SentenceTransformer(r"sentence-transformers/paraphrase-MiniLM-L6-v2")
output = model.encode(sentences=sentences,
show_progress_bar=True,
normalize_embeddings=True)
df_embedding = pd.DataFrame(output)
df_embedding
由于这一步骤非常重要,让我们逐点解释:
步骤1:为每一行创建包含完整客户/行信息的文本。我们还将其存储在一个Python列表中以备后用。请参见下图作为示例。
步骤2:这是调用transformer的步骤。为此,我们将使用存储在HuggingFace中的模型。该模型经过专门训练,可在句子级别执行嵌入,与Bert的模型不同,后者侧重于在标记和单词级别上进行编码。要调用模型,只需提供存储库地址,本例中为“sentence-transformers/paraphrase-MiniLM-L6-v2”。对于每个文本返回给我们的数值向量将被归一化,因为Kmeans模型对输入的尺度敏感。创建的向量长度为384。我们使用它们创建具有相同列数的数据框,见下图。
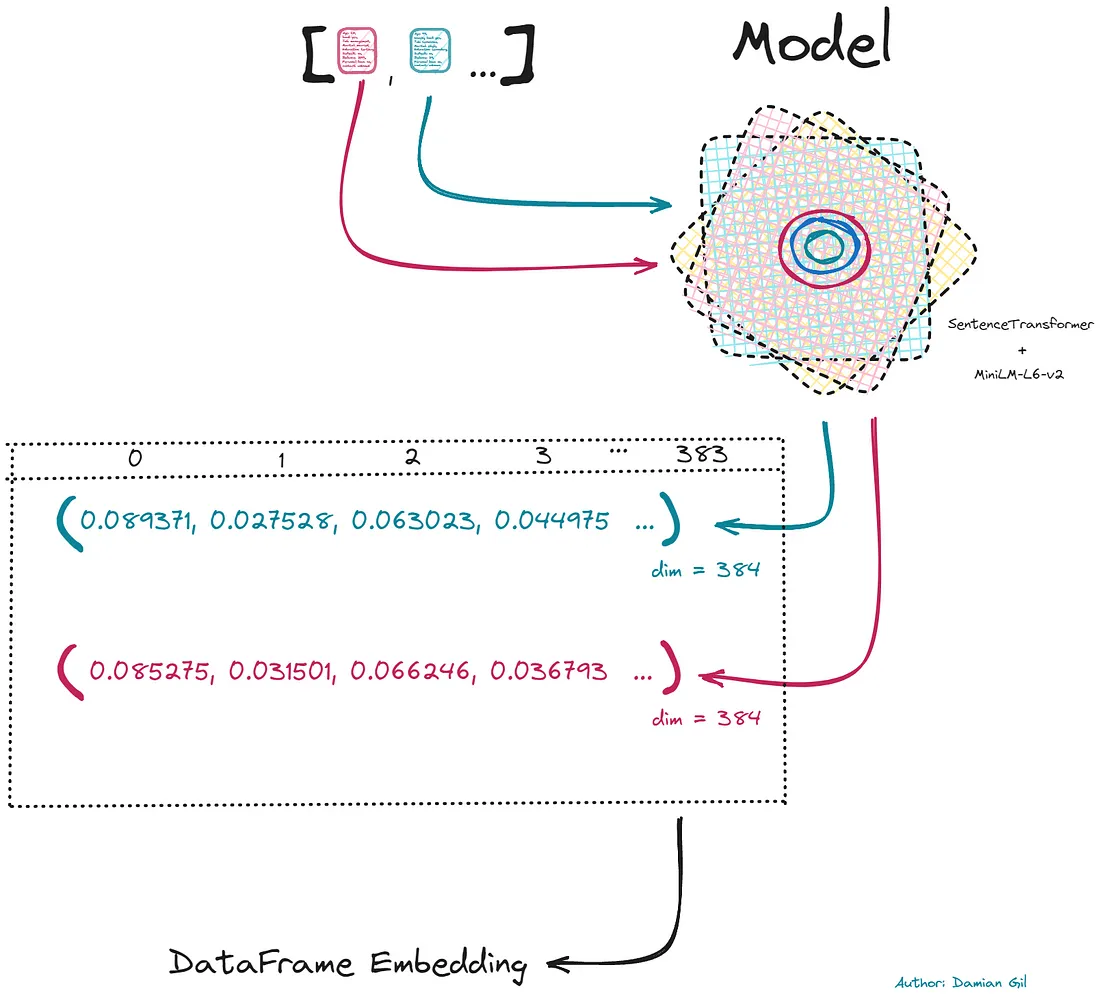
最后,我们从嵌入中获取数据框,这将成为我们Kmeans模型的输入。
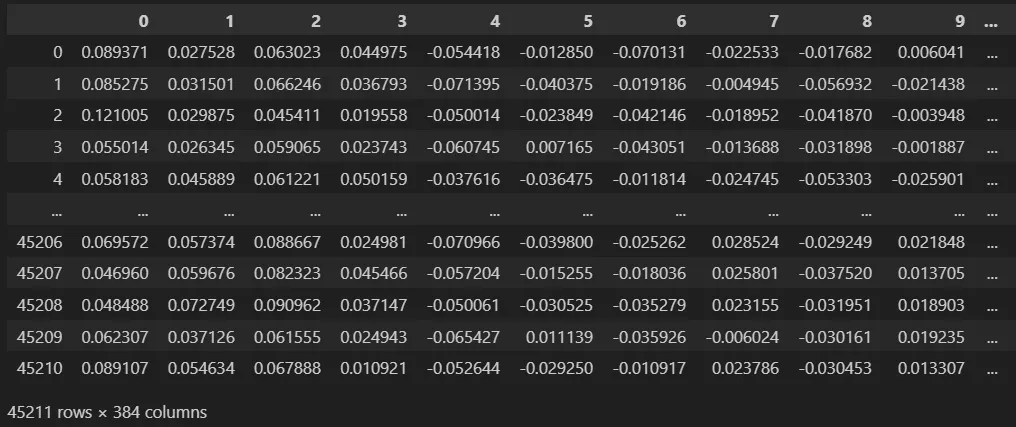
这一步是最有趣和重要的步骤之一,因为我们已经为我们将创建的Kmeans模型创建了输入。
创建和评估过程与上述相似。为了不使帖子过长,只会显示每个点的结果。
此外,应用词嵌入后得到的数据集已保存在一个名为embedding_train.csv的csv文件中。
# Normal Dataset
df = pd.read_csv("data/train.csv", sep = ";")
df = df.iloc[:, 0:8]
# Embedding Dataset
df_embedding = pd.read_csv("data/embedding_train.csv", sep = ",")
预处理
我们可以将嵌入视为预处理。
异常值
我们应用之前介绍的ECOD方法来检测异常值。我们创建一个不包含这些类型点的数据集。
df_embedding_no_out.shape -> (40690, 384)-> (40690, 384)
df_embedding_with_out.shape -> (45211, 384)
建模
首先,我们必须找出最佳聚类数量。为此,我们使用“Elbow Method”方法。
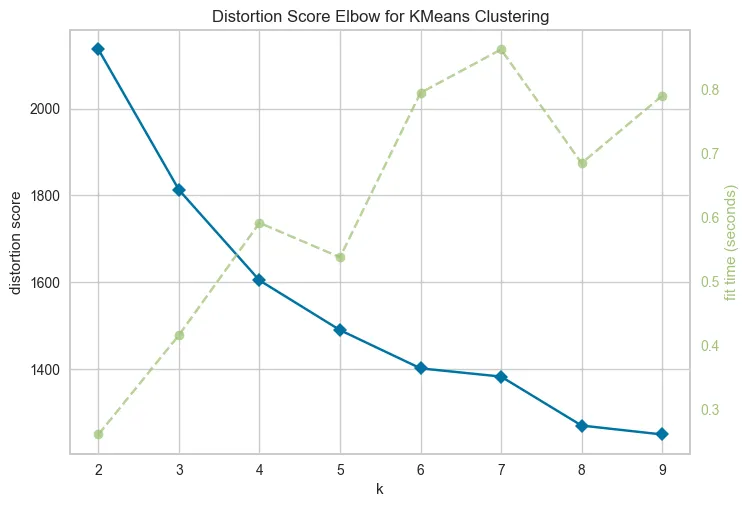
观看完图表后,我们选择k=5作为我们的聚类数量。
n_clusters = 5
clusters = KMeans(n_clusters=n_clusters, init = "k-means++").fit(df_embedding_no_out)"k-means++").fit(df_embedding_no_out)
print(clusters.inertia_)
clusters_predict = clusters.predict(df_embedding_no_out)
评估
下一步是使用k=5创建我们的Kmeans模型。接下来,我们可以获取一些度量指标,如下所示:
Davies bouldin score: 1.8095386826791042
Calinski Score: 6419.447089002081
Silhouette Score: 0.20360442824114108
由于观察到这些值与先前情况中获得的值非常相似。让我们研究通过主成分分析得到的表示结果:
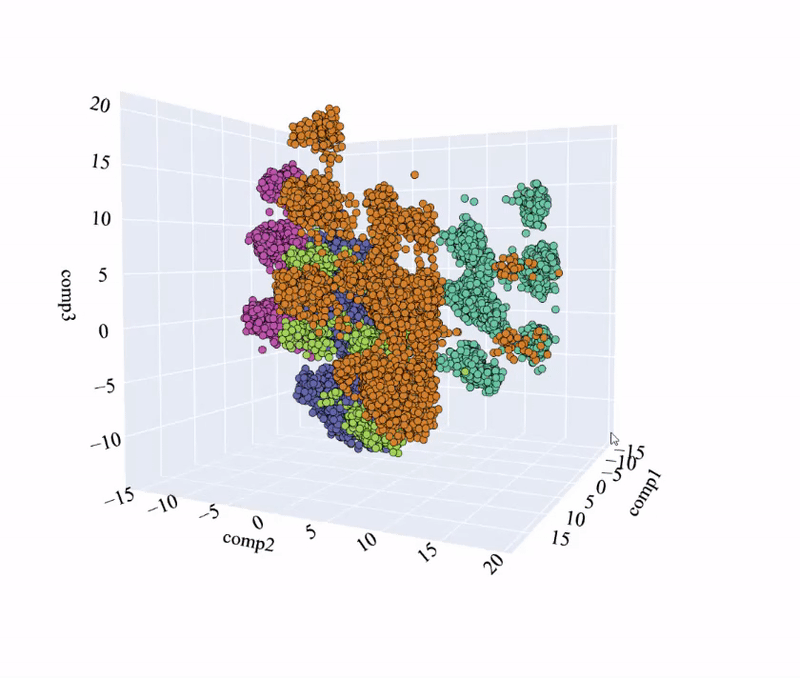
可以看到,与传统方法相比,聚类效果要好得多。这是一个好消息。让我们记住,在我们的主成分分析的前三个组成部分中,考虑到其中的变异性是重要的。从经验来看,当变异性约为50%(3D主成分分析)左右时,可以得出较为明确的结论。
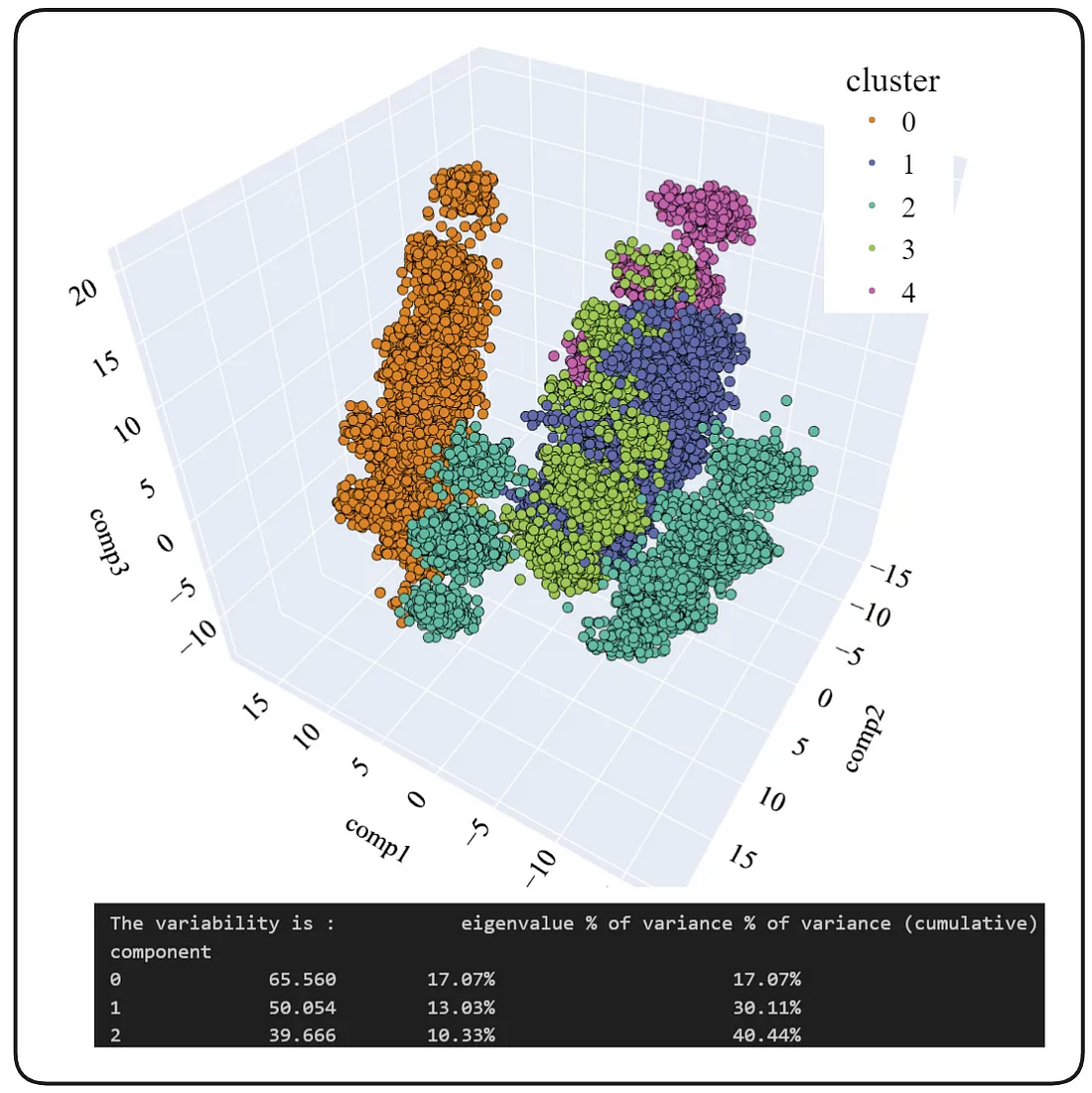
我们可以看到,四个组成部分的累积变化量为40.44%,这是可接受但并不理想。
通过修改三维表示中点的不透明度,我们可以直观地看到聚类有多紧密。这意味着当点在某个空间中聚集时,可以观察到一个黑点。如下图所示。
plot_pca_3d(df_pca_3d, title = "PCA Space", opacity=0.2, width_line = 0.1)"PCA Space", opacity=0.2, width_line = 0.1)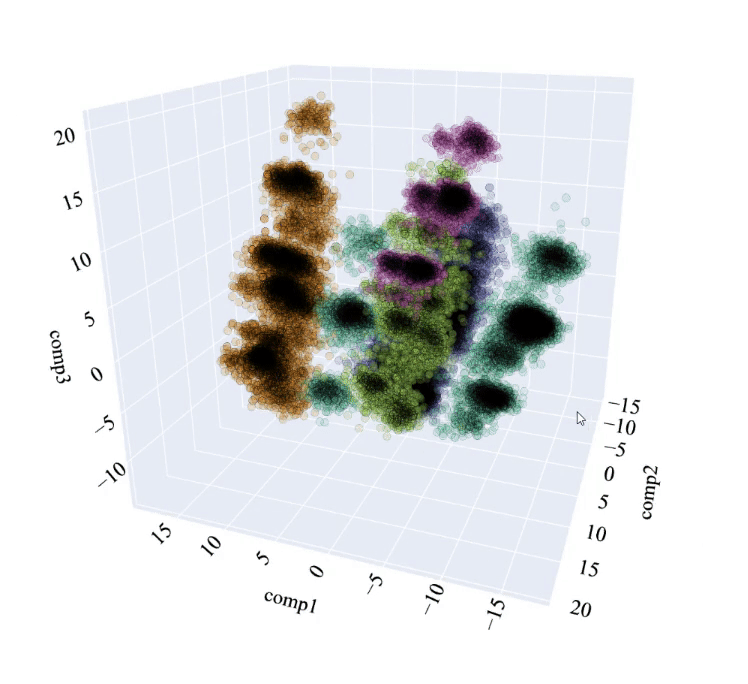
正如可以看到的那样,空间中存在着几个点,这些点在同一簇中聚集在一起。这表明它们与其他点之间有明显差异,模型能够很好地识别它们。
即便如此,可以看出,各个簇之间无法很好地区分。因此,我们进行了t-SNE分析,这是一种降低维度但保持空间结构的方法。
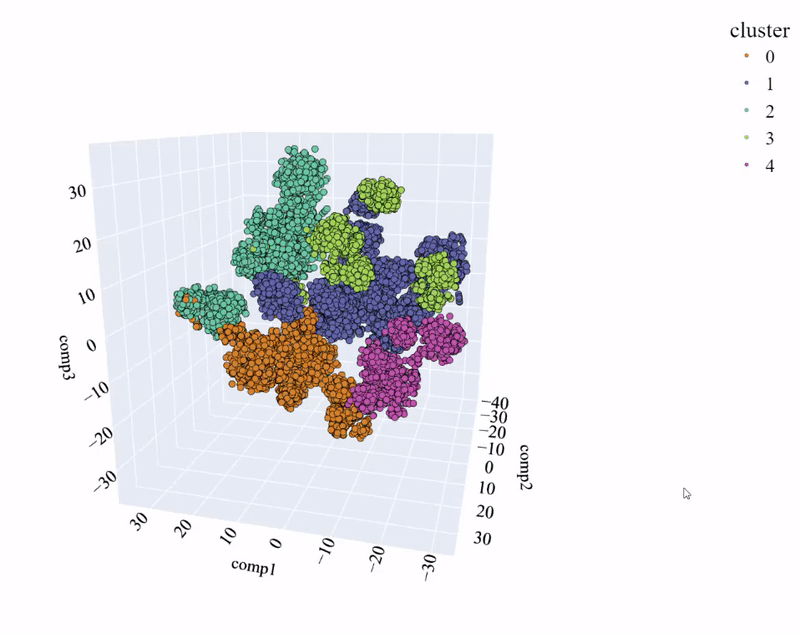
观察到明显的改进。聚类之间不会重叠,数据点之间有清晰的差异性。使用第二个降维方法获得的改进值得一提。让我们来看一个二维比较:
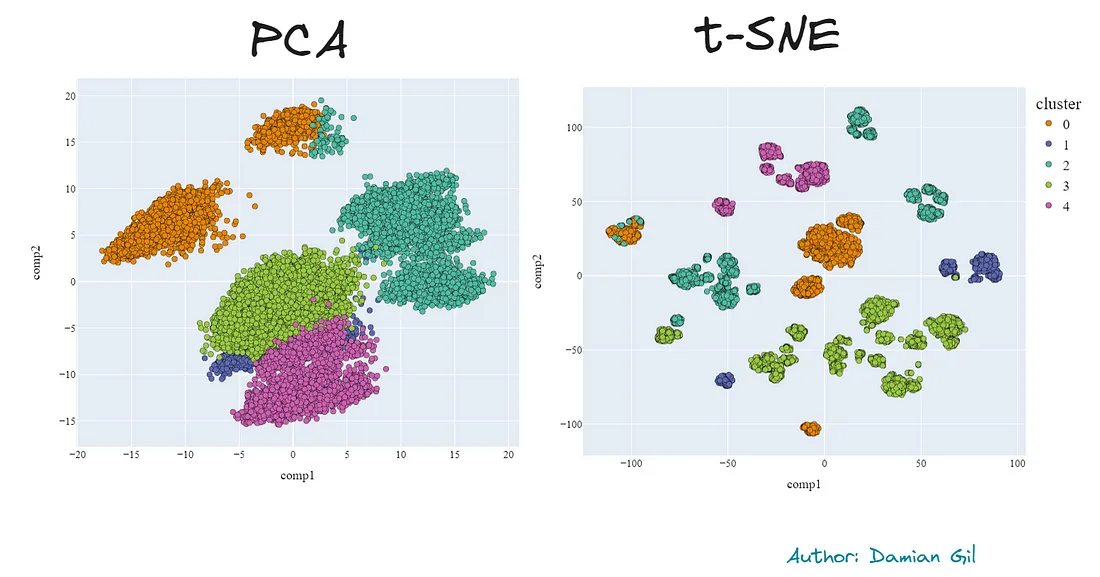
再次可以看到,t-SNE中的聚类比PCA中更分离且差异更大。此外,使用传统的Kmeans方法相比之下,两种方法的质量差异更小。
为了了解我们的Kmeans模型依赖于哪些变量,我们做了与之前相同的动作:创建一个分类模型(LGBMClassifier)并分析特征的重要性。
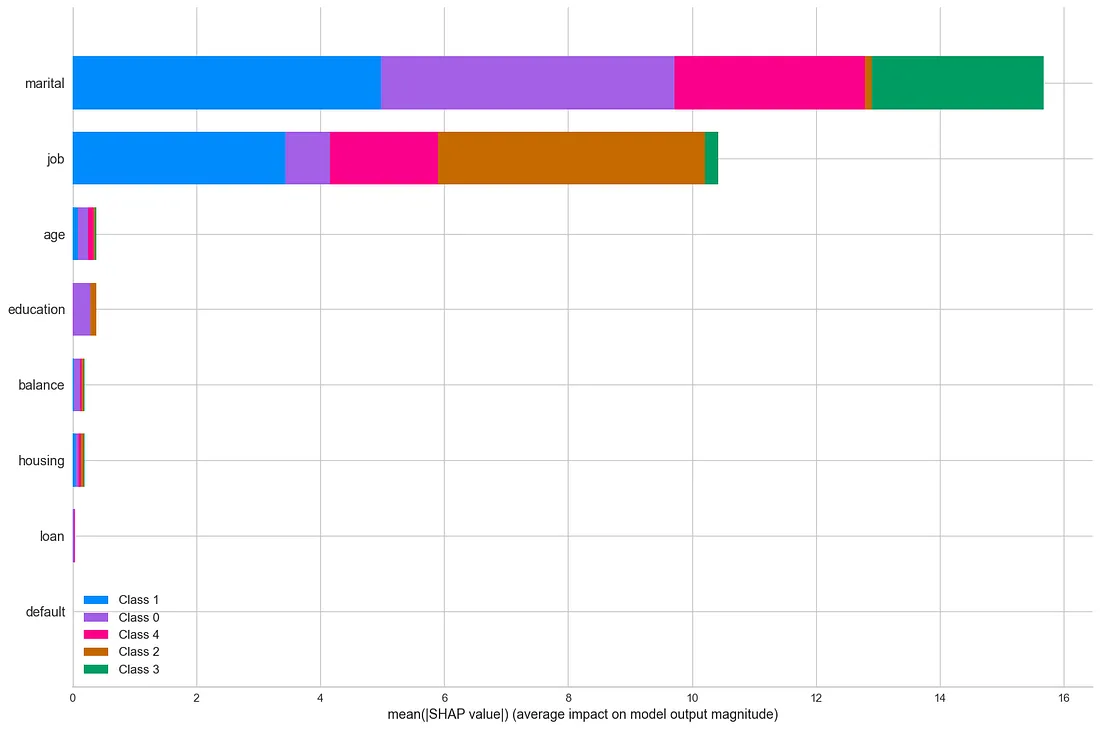
我们看到这个模型主要基于“婚姻”和“工作”变量。另一方面,我们看到还有一些变量没有提供太多信息。在一个真实的案例中,应该创建一个新版本的模型,去除这些信息较少的变量。
Kmeans+嵌入模型更加优化,因为它需要较少的变量就能够做出良好的预测。
我们结束了最具启示性和重要性的部分。
“经理和企业并不关心主成分分析(PCA)、t-分布随机邻居嵌入(t-SNE)或嵌入。他们想知道的是,对于他们的客户来说,主要的特征是什么。”
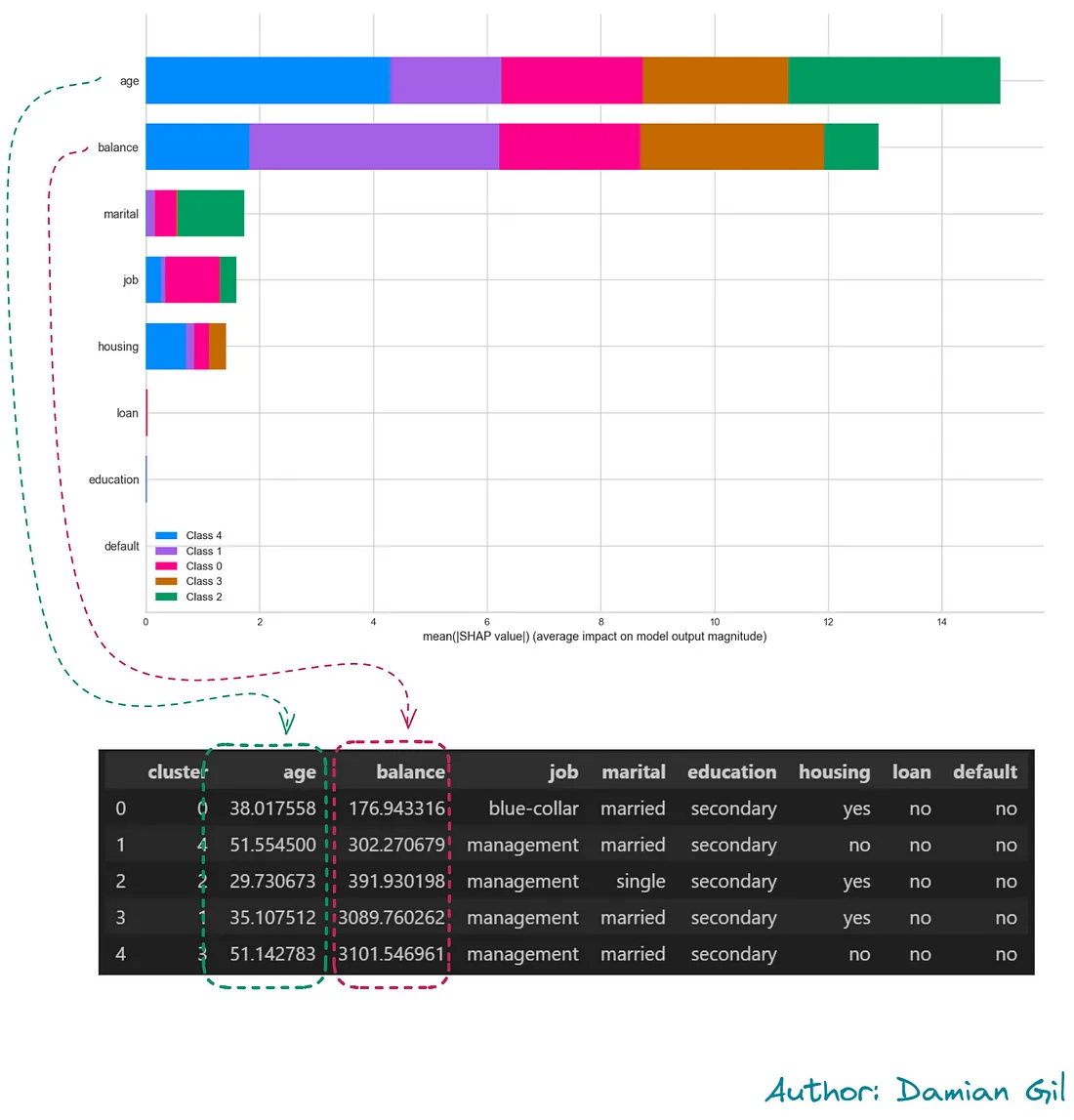
为此,我们创建了一个表格,其中包含了在每个聚类中我们能够找到的主导性的用户画像信息。
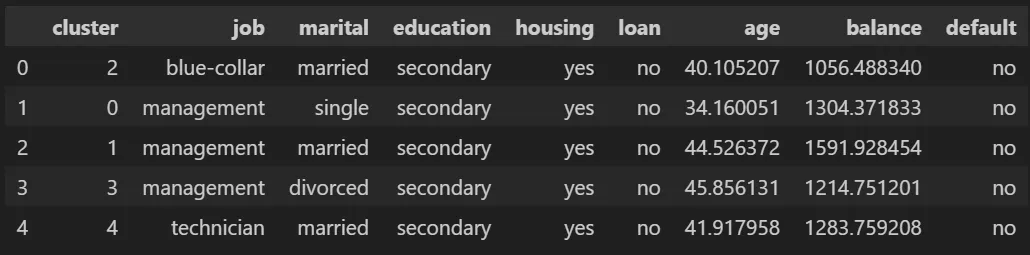
有一件非常有趣的事情发生:频率最高的职位聚集在“管理”一职的群体中,总共有3个群体。在这些群体中,我们发现了一种非常奇特的行为模式:单身经理年龄较小,已婚经理年龄较大,而离婚的经理年龄最大。另一方面,财务状况表现不同,单身人士的平均收入较离婚人士更高,而已婚人士的平均收入更高。以上述情况可以用以下图片概括:
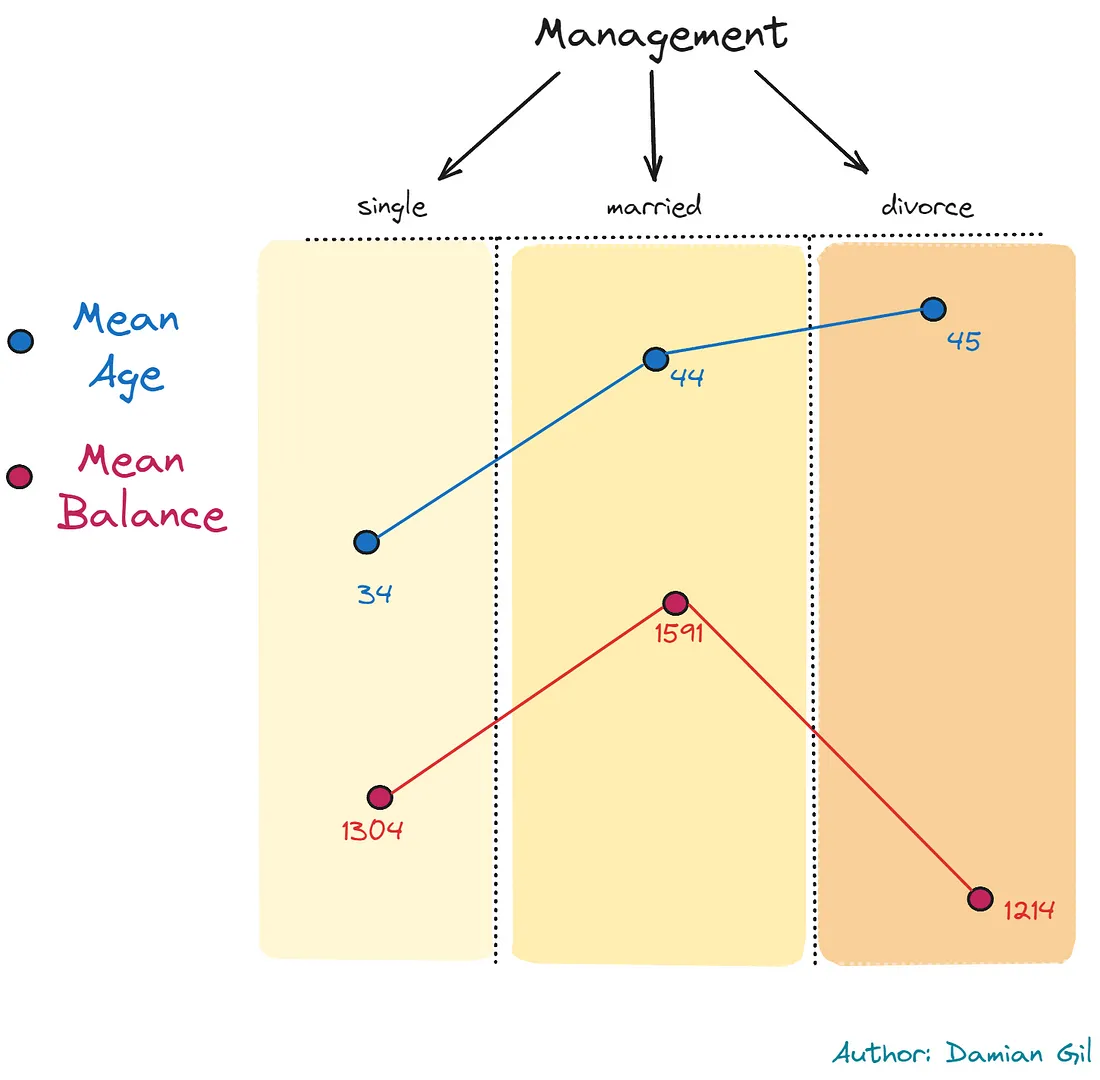
这个揭示与现实和社会方面是一致的。它还揭示了非常具体的客户画像。这就是数据科学的魅力。
结论
结论很明确:
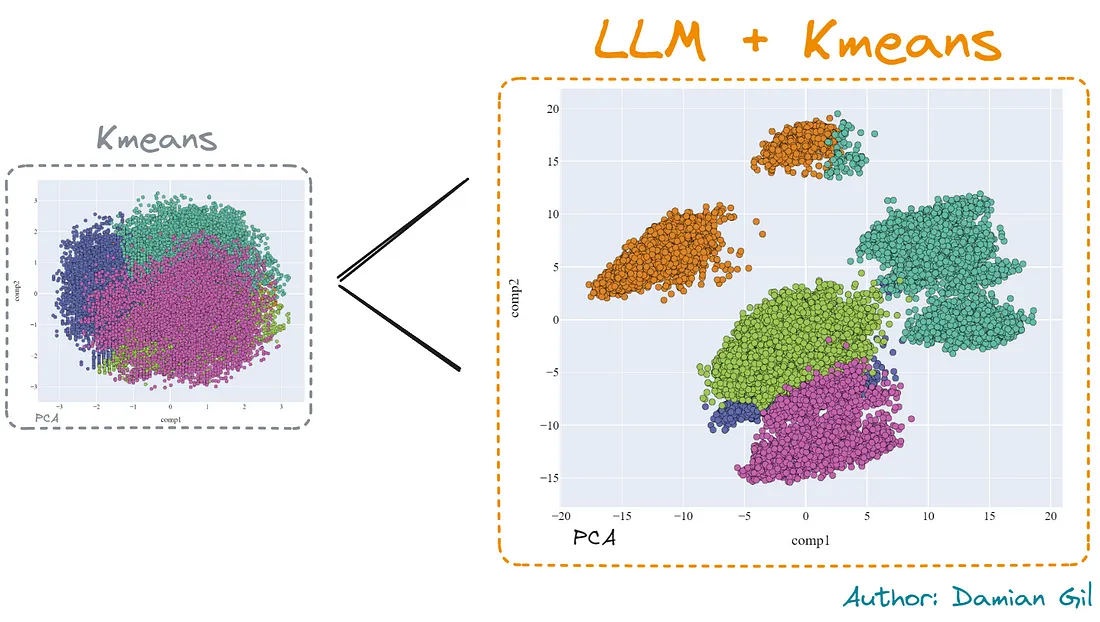
你必须拥有不同的工具,因为在真实的项目中,并不是所有的策略都适用,你必须拥有资源来增加价值。可以清晰地看到,借助LLM创建的模型脱颖而出。