ViTransUNet:利用混合架构彻底改变医学图像分割
介绍
随着先进神经网络架构的出现,医学成像领域经历了一场革命性的转变。在这些架构中,ViTransUNet脱颖而出,成为一项突破性的创新,它融合了视觉变换器(ViT)的能力和经典的U-Net模型。本文深入探讨了ViTransUNet的架构,它的运作机制,以及它在医学图像分割领域所产生的重大影响。
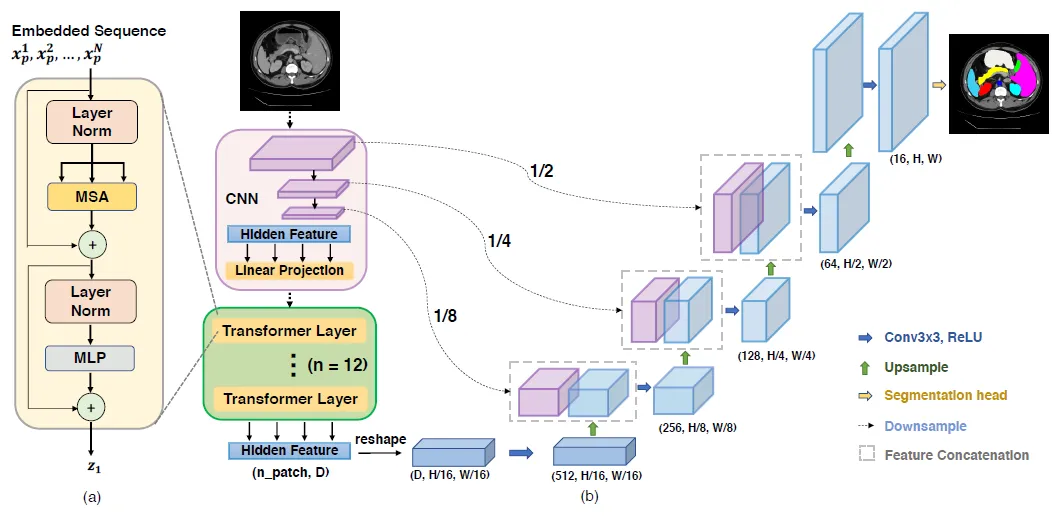
ViT 和 U-Net 在 ViTransUNet 中的融合
ViTransUNet的核心在于两种强大的神经网络架构:视觉变换器(ViT)和U-Net的融合。视觉变换器最初设计用于自然语言处理,现在已经灵活地被重新用于图像分析。通过将图像划分为补丁(patch)并顺序地处理这些补丁,ViT在识别图像数据中的复杂模式和内在关联方面表现出色。这种方法使得模型能够捕捉到图像的全局理解,这在识别包含的整体结构和模式方面至关重要。
相反,U-Net架构是医学影像处理中的中流砥柱,以其有效的分割能力而著称。它的结构特点是一个收缩路径以捕获上下文和一个对称扩展路径以实现精确的定位,这在详细和准确的分割任务中发挥了关键作用。U-Net在ViTransUNet中的整合带来了急需的局部特征提取和高分辨率分割能力。
ViTransUNet的操作机制
ViTransUNet的操作本质在于其能够将ViT的全局上下文分析与U-Net的局部精确性协同合作。模型首先将图像分割成多个补丁,通过变换器机制处理。这一阶段捕捉到了图像内更广泛的关系和模式。接下来,U-Net架构提炼这些发现,专注于局部细节并提高分割精确性。这种双重方法确保ViTransUNet能够处理具有细微细节的复杂医学图像,这一任务对于准确的医学分析至关重要。
医学成像及其他方面的应用
ViTransUNet的主要应用是在医学图像分割中,它已经显示出了显著的能力。其精确划分医学图像内的各种结构,如组织、器官或病理发现,使其在诊断成像如MRI、CT扫描和X射线中非常有价值。这种精确的分割有助于医疗专业人员在诊断、治疗规划和监测疾病进展中。
ViTransUNet是一个专为医学图像分割任务设计的专门神经网络架构。它结合了视觉变换器(ViT)和U-Net模型的优势,提供了一个强大的框架以精确识别和划分像MRI或CT扫描中的多种医学图像结构。
- 视觉变换器(ViT):这是ViTransUNet的一部分,它利用主要源自自然语言处理的变换器模型进行图像分析。ViT将图像分成补丁并依次处理这些补丁,使模型能够捕捉图像不同部分之间复杂的模式和关系。
- U-Net架构:U-Net是医学图像处理中流行的卷积神经网络架构。它以其在图像分割中的有效性而闻名,这是医学诊断中的关键任务。ViTransUNet中的U-Net组件有助于细化分割结果,使其更加准确和详细。
- 结合ViT和U-Net:ViTransUNet融合了这两种架构,利用ViT的全局接收场和自注意力机制与U-Net的局部特征提取和高分辨率分割能力。这种结合使ViTransUNet能够有效地分割复杂的医学图像,这对于准确的诊断和治疗规划至关重要。
- 医学成像应用:ViTransUNet可以应用于多种医学成像数据,例如MRI、CT扫描和X射线。它在需要精确分割组织、器官或异常的任务中特别有用,有助于医疗专业人员在诊断和治疗决策中。
- 优势:ViTransUNet的主要优势在于其能够捕捉图像中的全局和局部上下文信息,相较于传统方法,导致分割结果更准确。这在医学成像中尤为重要,精确划分解剖结构至关重要。
ViTransUNet代表了医学图像分析领域的重大进步,提供了改善图像分割任务的准确性和效率,这些任务在各种医学应用中至关重要。
此外,ViTransUNet的通用性不仅限于医学成像。其强大的架构可以适用于卫星图像分析、自动驾驶导航和生物研究等领域的其他分割任务,展现了其广泛的潜力。
优势和影响
ViTransUNet的主要优势是其全局和局部上下文分析的结合,导致更优越的分割结果。这种混合方法解决了传统分割方法的局限性,提供了更细致和详尽的分析。在医学领域,这意味着更准确的诊断和更明智的治疗决策,最终有助于改善病人的治疗结果。
代码
在Python中创建一个完整的ViTransUNet实现以及合成数据集是一项重大任务,但我可以引导你了解基本步骤并提供一个简化的示例以帮助你开始。ViTransUNet结合了视觉变换器(ViT)和U-Net架构进行医学图像分割,这需要理解深度学习模型和图像数据处理。
首先,确保你有必需的库。你可以使用pip安装它们:
pip install tensorflow torch torchvision
这个例子提供了一个基础的结构作为起点,但是要创建一个功能完备的ViTransUNet并配备一个全面的合成数据集,需要更加详细的代码、细致的调整和测试。如果你对深度学习或处理医学图像不熟悉,那么在尝试实施像ViTransUNet这样高级的模型之前,先熟悉一些更简单的模型可能会有所帮助。
import torch
import numpy as np
import tensorflow as tf
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from tensorflow.keras.layers import Conv2D, MaxPooling2D, Conv2DTranspose, concatenate
from torchvision.models import vit_b_16
# Load a pre-trained Vision Transformer model
vit_model = vit_b_16(pretrained=True)
def unet_model(input_size=(256, 256, 3)):
inputs = tf.keras.Input(input_size)
# Downscaling
conv1 = Conv2D(64, 3, activation='relu', padding='same')(inputs)
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)
# Further layers would be added here...
# Upscaling
up7 = Conv2DTranspose(64, 2, strides=(2, 2), padding='same')(pool1)
merge7 = concatenate([conv1, up7], axis=3)
conv7 = Conv2D(64, 3, activation='relu', padding='same')(merge7)
# Output layer
conv8 = Conv2D(1, 1, activation='sigmoid')(conv7)
model = tf.keras.Model(inputs=inputs, outputs=conv8)
return model
unet = unet_model()
def draw_circle(img_shape, radius, position):
"""Draw a circle on a numpy array."""
img = np.zeros(img_shape, dtype=np.float32)
rr, cc = np.ogrid[:img_shape[0], :img_shape[1]]
circle = (rr - position[0]) ** 2 + (cc - position[1]) ** 2 <= radius ** 2
img[circle] = 1
return img
def generate_synthetic_data(image_size=(256, 256), num_images=100):
images = np.zeros((num_images, *image_size, 3), dtype=np.float32)
masks = np.zeros((num_images, *image_size, 1), dtype=np.float32)
for i in range(num_images):
radius = np.random.randint(20, 50)
position = np.random.randint(50, 200, size=2)
circle_image = draw_circle(image_size, radius, position)
images[i, :, :, 0] = circle_image # Red channel
images[i, :, :, 1] = circle_image # Green channel
images[i, :, :, 2] = circle_image # Blue channel
masks[i, :, :, 0] = circle_image
return images, masks
synthetic_images, synthetic_masks = generate_synthetic_data()
# Splitting the dataset into training and testing sets
train_images, test_images, train_masks, test_masks = train_test_split(
synthetic_images, synthetic_masks, test_size=0.2, random_state=42
)
unet.compile(optimizer='adam', loss='binary_crossentropy', metrics=['accuracy'])
history = unet.fit(
train_images, train_masks,
validation_data=(test_images, test_masks),
epochs=5, # Use a larger number for real training
batch_size=32
)
test_loss, test_accuracy = unet.evaluate(test_images, test_masks)
print(f"Test Loss: {test_loss}, Test Accuracy: {test_accuracy}")
# Predicting masks on test images
predicted_masks = unet.predict(test_images)
# Displaying images, true masks, and predicted masks
def display_images(images, true_masks, predicted_masks, num_images=5):
plt.figure(figsize=(10, num_images * 3))
for i in range(num_images):
plt.subplot(num_images, 3, i * 3 + 1)
plt.imshow(images[i])
plt.title("Image")
plt.axis('off')
plt.subplot(num_images, 3, i * 3 + 2)
plt.imshow(true_masks[i, :, :, 0], cmap='gray')
plt.title("True Mask")
plt.axis('off')
plt.subplot(num_images, 3, i * 3 + 3)
plt.imshow(predicted_masks[i, :, :, 0], cmap='gray')
plt.title("Predicted Mask")
plt.axis('off')
plt.tight_layout()
plt.show()
display_images(test_images, test_masks, predicted_masks)
注意事项:
- 模型架构:本例采用了简化的U-Net模型。在实际应用中,将ViT整合进U-Net以构建ViTransUNet会更为复杂,需要对架构进行自定义修改。
- 超参数:周期数(epochs)、批量大小(batch size)以及其他超参数的选择应基于你的任务具体需求和数据的复杂程度。
- 数据增强:在真实世界场景中,尤其是医学图像,通常使用数据增强技术来提高模型的鲁棒性。
- 模型微调:对于像ViTransUNet这样复杂的模型来说,微调和针对变换器与U-Net整合所需的额外层是必要的。
- 验证和测试:为了做到更加严格的评估,可能需要采用更严格的验证技术,如k折交叉验证。
Epoch 1/5
3/3 [==============================] - 112s 34s/step - loss: 0.6813 - accuracy: 0.6136 - val_loss: 0.6609 - val_accuracy: 0.9899
Epoch 2/5
3/3 [==============================] - 106s 34s/step - loss: 0.6489 - accuracy: 0.9890 - val_loss: 0.6477 - val_accuracy: 0.9910
Epoch 3/5
3/3 [==============================] - 90s 26s/step - loss: 0.6348 - accuracy: 0.9902 - val_loss: 0.6276 - val_accuracy: 0.9924
Epoch 4/5
3/3 [==============================] - 74s 24s/step - loss: 0.6093 - accuracy: 0.9922 - val_loss: 0.5830 - val_accuracy: 0.9953
Epoch 5/5
3/3 [==============================] - 77s 25s/step - loss: 0.5576 - accuracy: 0.9959 - val_loss: 0.5110 - val_accuracy: 0.9993

请记住,训练深度学习模型,尤其是用于医学图像的模型,计算量非常大,需要仔细平衡各种超参数,并且需要大量数据才能获得有意义的结果。
结论
ViTransUNet 代表了神经网络架构在图像分析领域演进的一个重要里程碑。它将视觉变换器和U-Net的创新融合,为更准确、高效和详细的图像分割铺平了道路。这项技术不仅增强了医学成像的能力,也为需要精确和详细图像分析的各个其他领域开辟了新途径。随着我们继续探索人工智能在医疗保健中的潜力,ViTransUNet 作为杰出进步的一个见证,也象征着未来更伟大成就的前景。