Vision Transformer在医学图像分割中的实际应用和进展
本文的重点是视觉变换器(ViT)及其在语义分割问题上的实际应用。我将再次讨论磁共振图像上异常区域的分割任务。我已经使用 U-Net 解决了这一任务,并在此进行了讨论。此外,我还在这里介绍了在包含医学图像的自定义数据集上使用 ViT 进行图像分类的解决方案。
ViT 的基本特征如下:
- ViT 架构基于将图像表示为一组斑块。图像补丁是不重叠的图像块。每个区块都有一个嵌入向量,该向量最初由包含在该区块中的图像像素形成。
- 变换器编码器是 ViT 的主要部分,它根据图像的类别隶属关系来训练图像块之间的相似性。它包含一系列线性层、归一化层和激活层。
- 在大型数据集(如 ImageNet21K)上预先训练好的 ViT 模型可用于在自定义数据集上进行迁移学习,经过微调的模型表现出良好的性能。
U-Net 的基本特征如下:
- U-Net 由两部分组成: 编码器和解码器。编码器包含一系列特征提取和图像缩减块。解码器与编码器对称出现,并重建图像分辨率。
- U-Net 是 CNN 中用于医学图像语义分割的最佳架构之一。
在我的系统中,我使用 Hugging Face 的 Swin Transformer V2 作为分割编码器。Swin Transformer - 使用移位窗口的分层视觉变换器,包含 4 个阶段的编码器处理嵌入补丁。初始补丁大小为 4x4 像素。在每个编码器阶段,通过合并前一阶段较小补丁的嵌入,补丁的分辨率会提高两倍。这意味着,以补丁表示的图像空间分辨率在每个后续阶段都会降低两次。下图(来自 Hugging Face 文档)显示了 Swin Transformer 的高级架构:
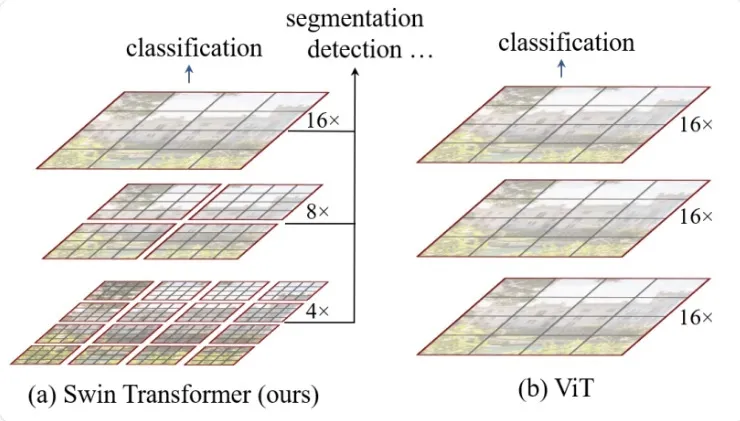
Swin Transformer 训练了多个分割模型,包括一个在 ImageNet21K 数据集(约 1,400 万张图片)上训练的大型模型。完整的分割管道包括编码器和解码器。Hugging Face 中的 Swin Transformer 编码器用于对自定义数据集进行微调。换句话说,我使用预先训练好的 Swin Transformer 大型模型作为编码器,并实现和训练我自己的解码器,从而在我的数据集上建立一个完整的语义分割系统。
Hugging Face的 Swin Transformer V2:深入内部
让我们使用以下代码块来看看 Hugging Face 的 Swin Transformer V2 模型:
安装:
!pip install torchvision
!pip install torchinfo
!pip install -q git+https://github.com/huggingface/transformers.git
导入:
from PIL import Image
from torchinfo import summary
import torch
安装 Google 驱动器(用于 Google Colab):
from google.colab import drive
drive.mount('/content/gdrive')
Cuda 设备设置:
device = torch.device('cuda') if torch.cuda.is_available() else torch.device('cpu')'cuda') if torch.cuda.is_available() else torch.device('cpu')加载大型预训练模型(在 ImageNet21K 上训练):
from transformers import AutoImageProcessor, Swinv2Model
image_processor = AutoImageProcessor.from_pretrained("microsoft/swinv2-large-patch4-window12-192-22k")
model = Swinv2Model.from_pretrained("microsoft/swinv2-large-patch4-window12-192-22k")
image_processor 定义了一组应用于输入图像的变换,输入图像最初为 PIL 图像:
ViTImageProcessor {
"_valid_processor_keys": ["_valid_processor_keys": [
"images",
"do_resize",
"size",
"resample",
"do_rescale",
"rescale_factor",
"do_normalize",
"image_mean",
"image_std",
"return_tensors",
"data_format",
"input_data_format"
],
"do_normalize": true,
"do_rescale": true,
"do_resize": true,
"image_mean": [
0.485,
0.456,
0.406
],
"image_processor_type": "ViTImageProcessor",
"image_std": [
0.229,
0.224,
0.225
],
"resample": 3,
"rescale_factor": 0.00392156862745098,
"size": {
"height": 192,
"width": 192
}
}输入的 PIL 图像转换为火炬张量,调整为 192x192 的图像分辨率,并进行归一化处理。
模型总结:
summary(model=model, input_size=(1, 3, 192, 192), col_names=['input_size', 'output_size', 'num_params', 'trainable'])1, 3, 192, 192), col_names=['input_size', 'output_size', 'num_params', 'trainable'])

这是一个大型模型,包含超过 1.95 亿个参数。
调用
model.eval()eval()
以查看模型包含的所有图层。
Swin Transformer V2 模型的组成部分如下:
- 补丁嵌入层:对于分辨率为 192x192 的输入图像,形成 2034=48*48 个大小为 4x4 的补丁。每个补丁形成一个长度为 192 的线性投影向量。
- 4 个编码器阶段。每个阶段都训练多头自注意。每个阶段以像素为单位的补丁大小增加两次(补丁嵌入合并)。每个阶段两次降低图片的像素分辨率。
- 对编码器输出进行归一化处理,生成编码器的最后隐藏状态。
- 对 last_hidden_state 张量进行平均池化,生成由类嵌入组成的池化输出向量。
让我们一步步看看它是如何工作的。
加载任何图像,并用图像处理器对其进行预处理:
import requests
url = "http://images.cocodataset.org/val2017/000000039769.jpg"
image = Image.open(requests.get(url, stream=True).raw)
inputs = image_processor(image, return_tensors="pt")
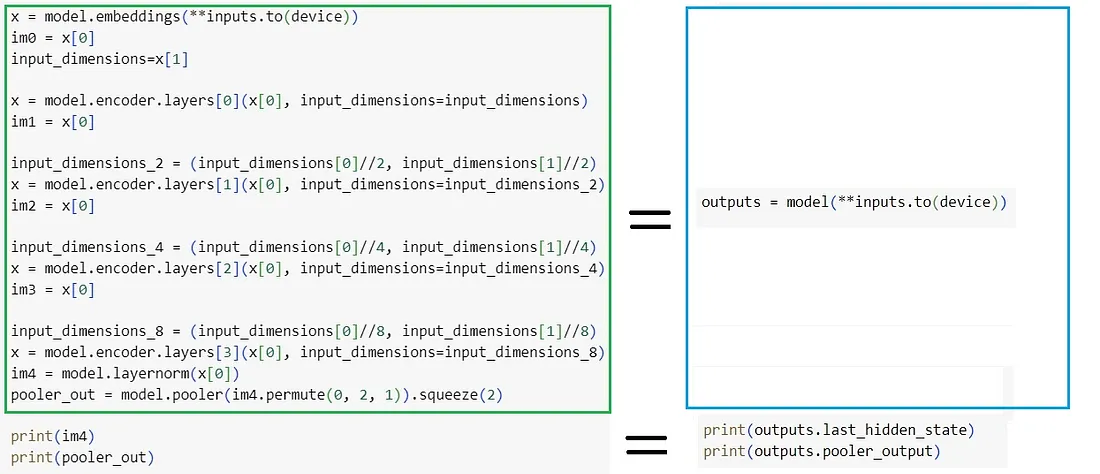
输入被发送到 Swin Transformer V2 模型。下图说明,按顺序调用 Swin Transformer V2 的各个部分(如左图所示)相当于调用整个模型(如右图所示):
查看每个编码器级的输出:
print(im0.shape)
print(im1.shape)
print(im2.shape)
print(im3.shape)
print(im4.shape)
我们可以看到以下形状:
im0 -> torch.Size([1, 2304, 192]) -> 2304=48*48 - 补丁数,192 - 补丁嵌入长度
im1 -> torch.Size([1, 576, 384]) -> 576=24*24 - 补丁数,384 - 补丁嵌入长度
im2 -> torch.Size([1, 144, 768]) -> 144=12*12 - 补丁数,768 - 补丁嵌入长度
im3 -> torch.Size([1, 36, 1536]) -> 36=6*6 - 补丁数,1536 - 补丁嵌入长度
im4 -> torch.Size([1, 36, 1536]) -> 36=6*6 - 补丁数,1536 - 补丁嵌入长度
这些来自 Swin Transformer V2 预训练大型模型的输出成为我的解码器模型的输入。我对解码器进行训练,以获得大脑 MRI 异常区域的分割掩码。下面的流程图显示了解码器的高级架构:
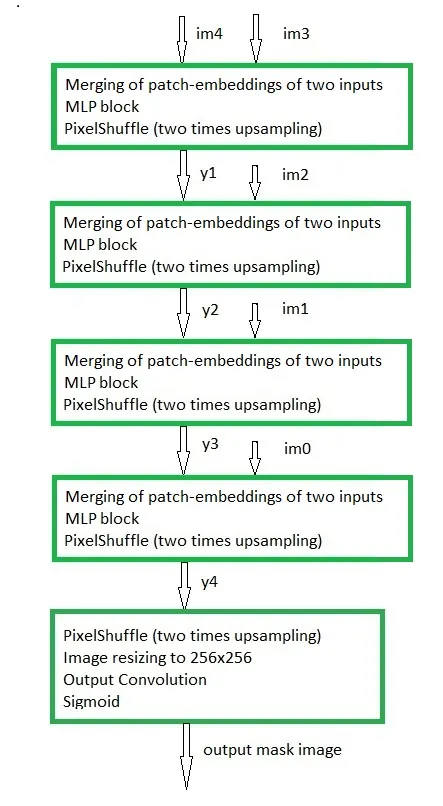
请注意,上述流程图最后一块中的 "图像大小调整为 256x256 "是普通解码器流程图中的自定义元素: 我使用 256x256 的图像分辨率来处理脑部 MRI 和分割掩码图像。
我使用 Swin Transformer V2 在大脑 MRI 上实现语义分割系统。变换器与 U-Net
让我们回到大脑核磁共振成像的语义分割任务。我使用的是 Kaggle 的大脑 MRI 数据集。该数据集包含 110 名患者的数据:一组带有脑切片的核磁共振成像,以及一组带有每位患者异常区域遮罩的相应图像。下图显示了 "脑切片图像 + 带掩膜图像 "对的示例:

在数据集中,每个人的 "大脑切片图像 + 掩膜图像 "对数从 20 对到 88 对不等。整个数据集包含 3935 对图像:2556 对为零掩码,1379 对为非零掩码的异常区域。
我使用 Swin Transformer V2 阶段(IM0、IM1、IM2、IM3、IM4 - 见图 1)的输出作为解码器的输入,实现并训练解码器模型。我使用 PyTorch 来实现该模型,并按照图 2 中的流程图来实现。
下面的代码显示了一幅图像的预处理过程,该图像最初是以 PIL 图像的形式从预训练的 Swin Transformer V2 模型阶段转换成 im0、im1、im2、im3、im4 张量。下面代码中的变量模型是在 ImageNet21K Swin Transformer V2 模型上加载的预训练模型(见上一节中的代码块):
img = <load PIL Image>
img = image_processor(images=img, return_tensors="pt")"pt")
x = model.embeddings(**img)
input_dimensions=x[1]
im0 = x[0].detach().squeeze()
x = model.encoder.layers[0](x[0], input_dimensions=input_dimensions)
im1 = x[0].detach().squeeze()
x = model.encoder.layers[1](x[0], input_dimensions=(input_dimensions[0]//2, input_dimensions[1]//2))
im2 = x[0].detach().squeeze()
x = model.encoder.layers[2](x[0], input_dimensions=(input_dimensions[0]//4, input_dimensions[1]//4))
im3 = x[0].detach().squeeze()
x = model.encoder.layers[3](x[0], input_dimensions=(input_dimensions[0]//8, input_dimensions[1]//8))
x = model.layernorm(x[0])
im4 = x.detach().squeeze()
注:我使用 squeeze() 来移除单个图像的批次尺寸,因为我认为它将被发送到 torch-DataLoader 中,而 torch-DataLoader 会为图像批次添加批次尺寸。
只有转换为 torch 张量和调整大小才会应用于遮罩图像。数据装载器中的 5 个输入张量批次将被发送到以下模型:
class Up_Linear(nn.Module):
def __init__(self, in_ch, size, coef=1):
super(Up_Linear, self).__init__()
self.shuffle = nn.PixelShuffle(upscale_factor=2)
n_ch = int(coef * in_ch)
self.ln = nn.Sequential(
nn.Linear(in_ch * 2, n_ch),
nn.ReLU(inplace=True),
nn.Linear(n_ch, in_ch * 2),
nn.ReLU(inplace=True),
)
self.size = size
def forward(self, x1, x2):
x = torch.cat((x1, x2), 2)
x = self.ln(x)
x = x.permute(0, 2, 1)
x = torch.reshape(x, (x.shape[0], x.shape[1], self.size, self.size))
x = self.shuffle(x)
x = torch.reshape(x, (x.shape[0], x.shape[1], self.size*self.size*4))
x = x.permute(0, 2, 1)
return x
class MRI_Seg(nn.Module):
def __init__(self):
super(MRI_Seg, self).__init__()
self.ups3 = Up_Linear(1536, 6, 1)
self.ups2 = Up_Linear(768, 12, 1)
self.ups1 = Up_Linear(384, 24, 2)
self.ups0 = Up_Linear(192, 48, 3)
self.shuffle = nn.PixelShuffle(upscale_factor=2)
self.out = nn.Sequential(
nn.Conv2d(24, 1, kernel_size=1, stride=1),
nn.Sigmoid()
)
def forward(self, x0, x1, x2, x3, x4):
x = self.ups3(x4, x3)
x = self.ups2(x, x2)
x = self.ups1(x, x1)
x = self.ups0(x, x0)
x = x.permute(0, 2, 1)
x = torch.reshape(x, (x.shape[0], x.shape[1], 96, 96))
x = self.shuffle(x)
x = transforms.Resize((256, 256))(x)
x = self.out(x)
return x
net = MRI_Seg().to(device)
该模型总结:
summary(model=net, input_size=[(1, 2304, 192), (1, 576, 384), (1, 144, 768), (1, 36, 1536), (1, 36, 1536)], col_names=['input_size', 'output_size', 'num_params', 'trainable'])1, 2304, 192), (1, 576, 384), (1, 144, 768), (1, 36, 1536), (1, 36, 1536)], col_names=['input_size', 'output_size', 'num_params', 'trainable'])
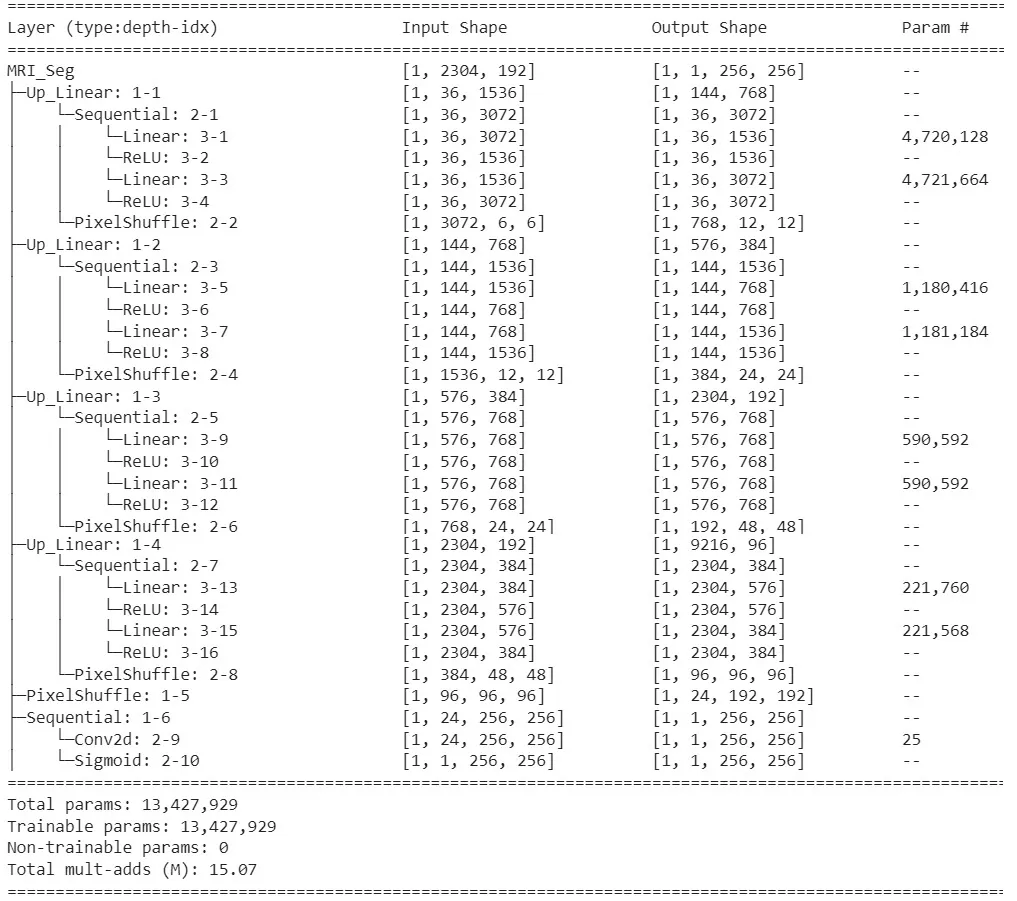
该模型包含 1,300 多万个可训练参数(如 U-Net 模型)。
我使用二元交叉熵损失函数来训练我的模型,以建立更接近标签(掩码)图像的掩码。我使用 Adam 优化器,学习率为 0.0001。我使用 IoU(Intersection over Union)和 Dice 指标作为质量度量:IoU = 1 和 Dice = 1 意味着理想的质量。请注意,在包括图片在内的所有结果中,我都使用了由训练有素的模型生成的带阈值应用的分割掩码:如果掩码像素值小于 0.5,则将其设置为 0,否则将其设置为 1。
下图显示了 U-Net 架构(在此获得并展示)和 MRI_Seg 模型(上图)的结果对比。对于这两个模型,我都选择了在测试集上显示最佳结果的检查点。在对 U-Net 和基于 Transformer 的模型进行性能评估时,我使用了从训练集中随机抽取的 550 个条目和测试集中包含的全部 394 个测试条目(两个模型使用了相同的训练集和测试集):
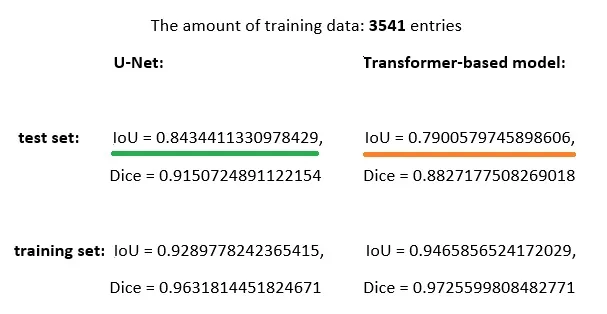
我们可以看到,基于变换器的分割模型的性能明显低于 U-Net 模型。我曾尝试改变解码器架构和可训练参数的数量,但并没有提高性能。
我将水平翻转(改变图像的左右两侧)应用于核磁共振成像和遮盖图像,创建了一个数据集,其图像数量比源数据集增加了一倍。现在,我使用这个增强数据集来训练基于 Transformer 的模型。下图显示了 U-Net 架构(已获得并在此展示)和 MRI_Seg 模型(上图)在增强数据集上的结果对比。对于这两个模型,我都选择了在测试集上显示最佳结果的检查点。在对 U-Net 和基于 Transformer 的模型进行性能评估时,我使用了从训练集中随机抽取的 550 个条目和包含在测试集中的所有 787 个测试条目(两个模型使用了相同的训练集和测试集):
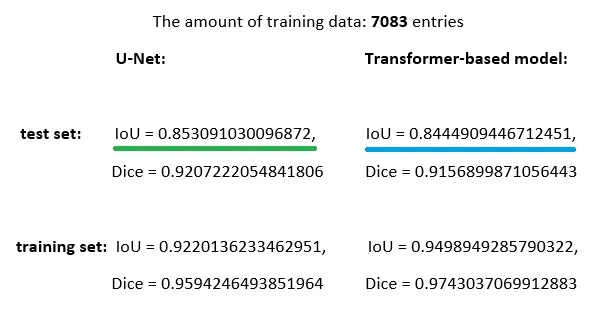
我们可以看到,在增强数据集上训练的基于 Transformer 的分割模型的性能有了显著提高,已接近 U-Net 模型的性能。
下图显示了基于 Transformer 训练的模型在测试图像上的工作结果。



结论
- Swin Transformer V2 高级架构允许使用 U-Net 模型的概念为语义分割系统实现自定义解码器。
- 自定义数据集的大小应足够大(约 10K)。在这种情况下,基于 Transformer 的语义分割系统性能良好。
- 基于 Transformer 的语义分割系统能够获得接近 U-Net 等最佳分割模型的性能。