如何使用GPT API从PDF出版物导出研究图表
介绍
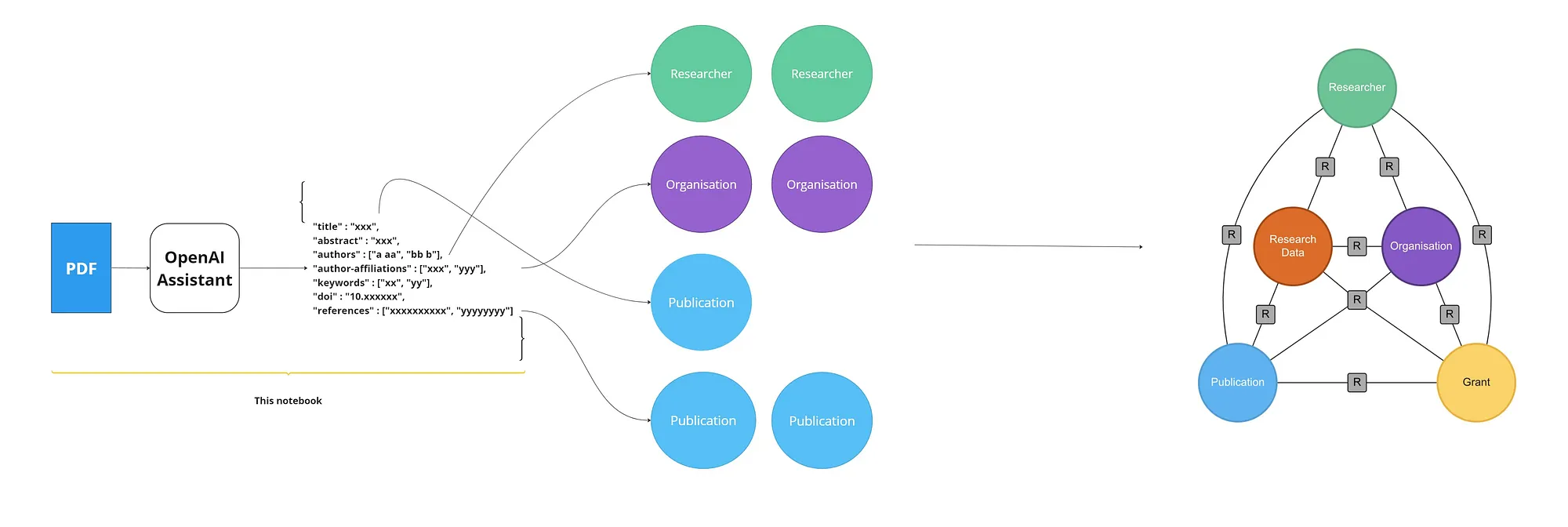
研究图是研究对象的结构化表示,它捕获有关实体的信息以及研究人员、组织、出版物、资助和研究数据之间的关系。目前,出版物以 PDF 文件形式提供,由于文本格式自由,因此很难解析 PDF 文件以提取结构化信息。在本文中,我们将尝试从出版物的 PDF 中创建研究图,方法是从文本中提取相关信息,并使用 OpenAI 将其组织成图结构。
OpenAI
在这项工作中,我们使用 OpenAI API 和 GPT 的新助手功能(目前处于测试阶段),将 PDF 文档转换为一组基于研究图模式的结构化 JSON 文件。
助手 API
Assistants API 允许你在应用程序中构建人工智能(AI)助理。助手可以根据预定准则使用模型、工具和信息回答用户的问题。这是一个测试版 API,目前正在积极开发中。使用助手 API,我们可以使用 OpenAI 托管的工具,如代码解释器和知识检索。在本文中,我们将重点介绍知识检索。
知识检索
有时,我们需要人工智能模型回答基于未知知识的查询,如用户提供的文档或敏感信息。我们可以使用助手 API 知识检索工具,用这些信息来增强模型。我们可以将文件上传到助手,它就会自动对文档进行分块,并创建和存储嵌入,从而在数据上实现矢量搜索。
举例说明
在我们的示例中,我们将向 OpenAI 助手和知识检索工具上传 PDF 格式的出版物文件,以获得给定出版物图模式的 JSON 输出。本示例中使用的出版物可通过以下链接访问。

步骤 1
读取存储出版物 PDF 的输入路径和存储 JSON 输出的输出路径。
import configparser
config = configparser.ConfigParser()
config.read('{}/config.ini'.format(current_path))
input_path = config['DEFAULT']['Input-Path']
output_path = config['DEFAULT']['Output-Path']
debug = config['DEFAULT']['Debug']
步骤 2
从输入路径中获取所有 PDF 文件。
onlyfiles = [f for f in os.listdir(input_path) if os.path.isfile(os.path.join(input_path, f))]
步骤 3
然后,我们需要初始化助手以使用知识检索工具。为此,我们需要在 API 中指定 “检索 ”工具的类型。我们还需要指定助手的指令和要使用的 OpenAI 模型。
my_file_ids = []
if client.files.list().data==[]:
for f in onlyfiles:
file = client.files.create(
file=open(input_path + f, "rb"),
purpose='assistants'
)
my_file_ids.append(file.id)
# Add the file to the assistant
assistant = client.beta.assistants.create(
instructions = "You are a publication database support chatbot. Use pdf files uploaded to best respond to user queries in JSON.",
model = "gpt-4-1106-preview",
tools = [{"type": "retrieval"}],
# Do not attach all files to the assistant, otherwise, it will mismatch the answers even though specify file ID in query messages.
# We will attach to each message instead
)
步骤 4
然后,我们指定需要从出版物文件中提取的信息,并将其作为用户查询传递给助手。在对助手的指令进行试验后,我们发现在每条用户信息中要求使用 JSON 格式能产生最一致的输出。
user_msgs = ["Print the title of this paper in JSON",
"Print the authors of this paper in JSON",
"Print the abstract section of the paper in JSON",
"Print the keywords of the paper in JSON",
"Print the DOI number of the paper in JSON",
"Print the author affiliations of the paper in JSON",
"Print the reference section of the paper in JSON"]
步骤 5
下一步是将查询传递给助手以生成输出。我们需要为每个用户查询创建一个单独的线程对象,其中包含作为用户消息的查询。然后,我们运行线程并获取助手的答案。
all_results = []
for i in my_file_ids:
print('\n#####')
# the JSON result it can extract and parse, hopefully
file_result = {}
for q in user_msgs:
# create thread, user message and run objects for each query
thread = client.beta.threads.create()
msg = client.beta.threads.messages.create(
thread_id=thread.id,
role="user",
content=q,
file_ids=[i] # specify the file/publication we want to extract from
)
print('\n',q)
run = client.beta.threads.runs.create(
thread_id=thread.id,
assistant_id=assistant.id,
additional_instructions="If answer cannot be found, print 'False'" # not very useful at the time of this presentation
)
# checking run status by retrieving updated object each time
while run.status in ["queued",'in_progress']:
print(run.status)
time.sleep(5)
run = client.beta.threads.runs.retrieve(
thread_id=thread.id,
run_id=run.id
)
# usually a rate limit error
if run.status=='failed': logging.info("Run failed: ", run)
if run.status=='completed':
print("<Complete>")
# extract updated message object, this includes user messages
messages = client.beta.threads.messages.list(
thread_id=thread.id
)
for m in messages:
if m.role=='assistant':
value = m.content[0].text.value # get the text response
if "json" not in value:
if value=='False':logging.info("No answer found for ", str(q))
else:
logging.info("Not JSON output, maybe no answer found in the file or model is outdated: ", str(value))
else:
# clean the response and try to parse as json
value = value.split("```")[1].split('json')[-1].strip()
try:
d = json.loads(value)
file_result.update(d)
print(d)
except Exception as e:
logging.info(f"Query {q} \nFailed to parse string to JSON: ", str(e))
print(f"Query {q} \nFailed to parse string to JSON: ", str(e))
all_results.append(file_result)
上述出版文件生成的 JSON 输出为:
[{'title': 'Dodes (diagnostic nodes) for Guideline Manipulation',
'authors': [{'name': 'PM Putora',
'affiliation': 'Department of Radiation-Oncology, Kantonsspital St. Gallen, St. Gallen, Switzerland'},
{'name': 'M Blattner',
'affiliation': 'Laboratory for Web Science, Zürich, Switzerland'},
{'name': 'A Papachristofilou',
'affiliation': 'Department of Radiation Oncology, University Hospital Basel, Basel, Switzerland'},
{'name': 'F Mariotti',
'affiliation': 'Laboratory for Web Science, Zürich, Switzerland'},
{'name': 'B Paoli',
'affiliation': 'Laboratory for Web Science, Zürich, Switzerland'},
{'name': 'L Plasswilma',
'affiliation': 'Department of Radiation-Oncology, Kantonsspital St. Gallen, St. Gallen, Switzerland'}],
'Abstract': {'Background': 'Treatment recommendations (guidelines) are commonly represented in text form. Based on parameters (questions) recommendations are defined (answers).',
'Objectives': 'To improve handling, alternative forms of representation are required.',
'Methods': 'The concept of Dodes (diagnostic nodes) has been developed. Dodes contain answers and questions. Dodes are based on linked nodes and additionally contain descriptive information and recommendations. Dodes are organized hierarchically into Dode trees. Dode categories must be defined to prevent redundancy.',
'Results': 'A centralized and neutral Dode database can provide standardization which is a requirement for the comparison of recommendations. Centralized administration of Dode categories can provide information about diagnostic criteria (Dode categories) underutilized in existing recommendations (Dode trees).',
'Conclusions': 'Representing clinical recommendations in Dode trees improves their manageability handling and updateability.'},
'Keywords': ['dodes',
'ontology',
'semantic web',
'guidelines',
'recommendations',
'linked nodes'],
'DOI': '10.5166/jroi-2-1-6',
'references': [{'ref_number': '[1]',
'authors': 'Mohler J Bahnson RR Boston B et al.',
'title': 'NCCN clinical practice guidelines in oncology: prostate cancer.',
'source': 'J Natl Compr Canc Netw.',
'year': '2010 Feb',
'volume_issue_pages': '8(2):162-200'},
{'ref_number': '[2]',
'authors': 'Heidenreich A Aus G Bolla M et al.',
'title': 'EAU guidelines on prostate cancer.',
'source': 'Eur Urol.',
'year': '2008 Jan',
'volume_issue_pages': '53(1):68-80',
'notes': 'Epub 2007 Sep 19. Review.'},
{'ref_number': '[3]',
'authors': 'Fairchild A Barnes E Ghosh S et al.',
'title': 'International patterns of practice in palliative radiotherapy for painful bone metastases: evidence-based practice?',
'source': 'Int J Radiat Oncol Biol Phys.',
'year': '2009 Dec 1',
'volume_issue_pages': '75(5):1501-10',
'notes': 'Epub 2009 May 21.'},
{'ref_number': '[4]',
'authors': 'Lawrentschuk N Daljeet N Ma C et al.',
'title': "Prostate-specific antigen test result interpretation when combined with risk factors for recommendation of biopsy: a survey of urologist's practice patterns.",
'source': 'Int Urol Nephrol.',
'year': '2010 Jun 12',
'notes': 'Epub ahead of print'},
{'ref_number': '[5]',
'authors': 'Parmelli E Papini D Moja L et al.',
'title': 'Updating clinical recommendations for breast colorectal and lung cancer treatments: an opportunity to improve methodology and clinical relevance.',
'source': 'Ann Oncol.',
'year': '2010 Jul 19',
'notes': 'Epub ahead of print'},
{'ref_number': '[6]',
'authors': 'Ahn HS Lee HJ Hahn S et al.',
'title': 'Evaluation of the Seventh American Joint Committee on Cancer/International Union Against Cancer Classification of gastric adenocarcinoma in comparison with the sixth classification.',
'source': 'Cancer.',
'year': '2010 Aug 24',
'notes': 'Epub ahead of print'},
{'ref_number': '[7]',
'authors': 'Rami-Porta R Goldstraw P.',
'title': 'Strength and weakness of the new TNM classification for lung cancer.',
'source': 'Eur Respir J.',
'year': '2010 Aug',
'volume_issue_pages': '36(2):237-9'},
{'ref_number': '[8]',
'authors': 'Sinn HP Helmchen B Wittekind CH.',
'title': 'TNM classification of breast cancer: Changes and comments on the 7th edition.',
'source': 'Pathologe.',
'year': '2010 Aug 15',
'notes': 'Epub ahead of print'},
{'ref_number': '[9]',
'authors': 'Paleri V Mehanna H Wight RG.',
'title': "TNM classification of malignant tumours 7th edition: what's new for head and neck?",
'source': 'Clin Otolaryngol.',
'year': '2010 Aug',
'volume_issue_pages': '35(4):270-2'},
{'ref_number': '[10]',
'authors': 'Guarino N.',
'title': 'Formal Ontology and Information Systems',
'source': '1998 IOS Press'},
{'ref_number': '[11]',
'authors': 'Uschold M Gruniger M.',
'title': 'Ontologies: Principles Methods and Applications.',
'source': 'Knowledge Engineering Review',
'year': '1996',
'volume_issue_pages': '11(2)'},
{'ref_number': '[12]',
'authors': 'Aho A Garey M Ullman J.',
'title': 'The Transitive Reduction of a Directed Graph.',
'source': 'SIAM Journal on Computing',
'year': '1972',
'volume_issue_pages': '1(2): 131–137'},
{'ref_number': '[13]',
'authors': 'Tai K',
'title': 'The tree-to-tree correction problem.',
'source': 'Journal of the Association for Computing Machinery (JACM)',
'year': '1979',
'volume_issue_pages': '26(3):422-433'}]}]步骤 6
文件对象和助手对象需要清理,因为它们在 “检索 ”模式下需要花钱。此外,这也是一种良好的编码实践。
for f in client.files.list().data:
client.files.delete(f.id)
# Retrieve and delete running assistants
my_assistants = client.beta.assistants.list(
order="desc",
)
for a in my_assistants.data:
response = client.beta.assistants.delete(a.id)
print(response)
步骤 7
下一步是使用 Python Networkx 软件包生成图形可视化。
import networkx as nx
import matplotlib.pyplot as plt
G = nx.DiGraph()
node_colors = []
key = "jroi/" + all_results[0]['title']
G.add_nodes_from([(all_results[0]['title'], {'doi': all_results[0]['DOI'], 'title': all_results[0]['title'], 'source': 'jroi', 'key': key})])
node_colors.append('#4ba9dc')
for author in all_results[0]['authors']:
key = "jroi/" + author['name']
G.add_nodes_from([(author['name'], {'key': key, 'local_id': author['name'], 'full_name': author['name'], 'source': 'jroi'})])
G.add_edge(all_results[0]['title'], author['name'])
node_colors.append('#63cc9e')
for reference in all_results[0]['references']:
key = "jroi/" + reference['title']
G.add_nodes_from([(reference['title'].split('.')[0][:25] + '...', {'title': reference['title'], 'source': 'jroi', 'key': key})])
G.add_edge(all_results[0]['title'], reference['title'].split('.')[0][:25] + '...')
node_colors.append('#4ba9dc')
pos = nx.spring_layout(G)
labels = nx.get_edge_attributes(G, 'label')
nx.draw(G, pos, with_labels=True, node_size=1000, node_color=node_colors, font_size=7, font_color='black')
nx.draw_networkx_edge_labels(G, pos, edge_labels=labels)
plt.savefig("graph_image.png")
plt.show()
可视化图表如下:
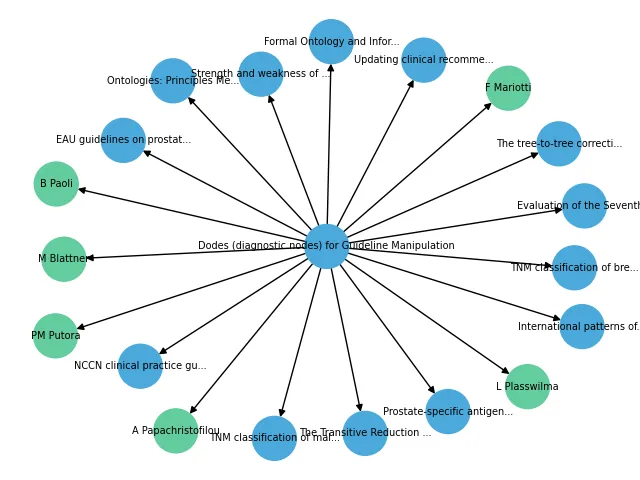
注:请注意,OpenAI 生成的输出结构可能因执行方式不同而不同。因此,你可能需要根据该结构更新上述代码。
结论
总之,利用 GPT API 从 PDF 出版物中提取研究图为研究人员和数据分析师提供了一个强大而高效的解决方案。该工作流程简化了将 PDF 出版物转换为结构化、可访问的研究图表的过程。但是,我们也必须仔细关注大型语言模型 (LLM) 生成的回复的不一致性。随着时间的推移,通过定期更新和改进提取模型,可以进一步提高准确性和相关性。