PrimeKG:医学和医疗保健知识图谱
介绍
万物相连。因此,用图对世界建模是一个自然的选择。例如,MONDO 疾病本体- 一个由数百万个实体(从基因到疾病)组成的知识图谱;或基因本体 - 描述分子功能、细胞成分和生物过程。此外,还有一些数据库,如疾病基因网络(DisGeNet)--探索基因与疾病的关联;Orphanet--罕见病百科全书。如图所示,这些医学和保健知识是零散的。PrimeKG试图...
......整合了 20 个高质量资源,描述了 17,080 种疾病与 4,050,249 种关系,代表了十大生物学尺度,包括与疾病相关的蛋白质扰动、生物学过程和途径、解剖学和表型尺度,以及全部已批准药物及其治疗作用,极大地扩展了以往在以疾病为基础的知识图谱方面所做的努力。
准备和装载
准备
- 请点击 PrimeKG 原始数据链接。下载 "edges.csv"(368.6 MB)和 "nodes.csv"(7.5 MB),为什么?→还有其他文件,例如 "kg.csv",它包含完整的图形(每行的格式为node1-relation-node2)。但是用单独的节点和边文件导入图形是最快捷、最有效的方法。
- 下载并安装开放式 JDK。我经常使用 Eclipse Temurin。选择版本 17 (LTS) 或 21 (LTS)。安装完成后,检查 JAVA_HOME 环境变量是否指向正确的 JDK 位置。为什么?→以后我们需要 JDK 才能在 Neo4j 中运行 "neo4j-admin.bat"。
- 使用 Notepad++ 将 edges.csv 的标题改为 :TYPE,display_relation,:START_ID,:END_ID。将 "nodes.csv "的页眉改为 node_index:ID,node_id,:LABEL,node_name,node_source。为什么?→Neo4j 导入工具要求 CSV 文件的标题遵循某些准则,例如哪一列是 ID。
- 使用 Notepad++ 在 "nodes.csv "中搜索并替换"/"为"__"。为什么? → Neo4j 节点名称必须遵循某些限制。
- 下载并安装 Anaconda,或至少安装带有 Pandas 库的 Python。为什么?→不幸的是,我发现 "edges.csv "包含双重边缘,这意味着我们有一行 CSV 记录 "节点 A 指向节点 B,关系为 X",而另一行记录 "节点 B 指向节点 A,关系为 X"。我们打算在 Neo4j 中构建一个无向图。因此,我们必须删除重复的边。我只知道如何用 Python/Pandas 来做这件事!
import pandas as pd
df = pd.read_csv(r"edges.csv", header="infer", sep=",", encoding="utf-8", dtype=str, keep_default_na=False)
group = df[[":START_ID", ":END_ID", ":TYPE", "display_relation"]].agg(frozenset, axis=1)
df_result = df.groupby(group).first()
df_result.to_csv(r"edges2.csv", sep=',', encoding='utf-8', index=False)
正在加载
- 打开 Neo4j Desktop。创建新项目。添加 "本地 DBMS"。查找三点:点击它,然后点击 "打开文件夹",再点击 "DBMS"。
- Windows 资源管理器会打开 DBMS 所在的位置。在我的例子中,是 C:\Users\[用户名]\.Neo4jDesktop\relate-data\dbmss\dbms-886ac993-25d9-493a-922b-2d41e29b6446。
- 在这里打开 CMD。现在使用下面的命令。记住要适当更改 [path]。
.\bin\neo4j-admin database import full primekg --nodes="D:\[path]\nodes.csv" --relationships="D:\[path]\edges2.csv" --trim-strings=true"D:\[path]\nodes.csv" --relationships="D:\[path]\edges2.csv" --trim-strings=true
- 在 Neo4j Desktop 中,根据已创建的 DBMS 点击 "Start(开始)"按钮。然后点击 "Open(打开)",打开 Neo4j 浏览器。
- 创建数据库 "primekg"。然后改用该数据库作为活动数据库。(这些都是 Cypher 查询)
CREATE DATABASE primekg
:use primekg
在 Neo4j 浏览器中,输入命令来验证是否一切正常:
MATCH (n) RETURN COUNT(n)
命令应返回 129375。这就是图中的节点数。然后使用命令进一步测试:
MATCH ()-[r]-() RETURN COUNT(DISTINCT r)
它应该返回 4050249。
可视化与探索
可视化
要获取数据库模式,我们可以使用 Neo4j 浏览器中的 CALL db.schema.visualization()。一个可视化图表就会显示出来:
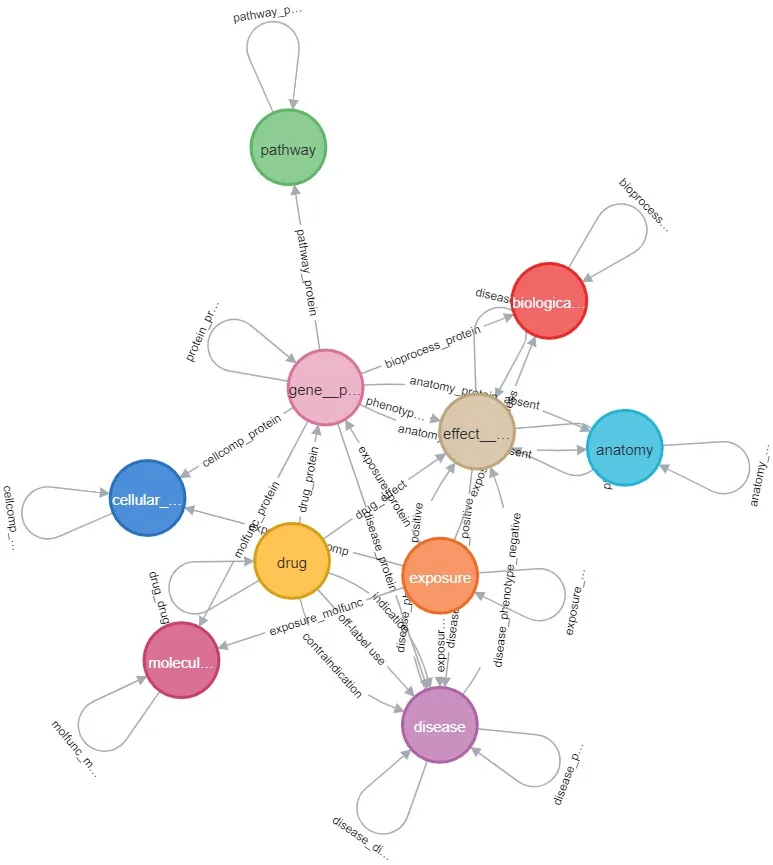
探索
要获取所有节点类型,请使用 CALL db.labels()。结果:
╒════════════════════╕
│label │
╞════════════════════╡
│"anatomy" │"anatomy" │
├────────────────────┤
│"gene__protein" │
├────────────────────┤
│"disease" │
├────────────────────┤
│"effect__phenotype" │
├────────────────────┤
│"drug" │
├────────────────────┤
│"biological_process"│
├────────────────────┤
│"molecular_function"│
├────────────────────┤
│"cellular_component"│
├────────────────────┤
│"exposure" │
├────────────────────┤
│"pathway" │
└────────────────────┘
在论文的第 5 页,我们可以看到节点列表,它们是匹配的!
使用 CALL db.relationshipTypes() 获取所有关系类型。结果
╒════════════════════════════╕
│relationshipType │
╞════════════════════════════╡
│"protein_protein" │"protein_protein" │
├────────────────────────────┤
│"anatomy_protein_present" │
├────────────────────────────┤
│"molfunc_protein" │
├────────────────────────────┤
│"cellcomp_protein" │
├────────────────────────────┤
│"drug_effect" │
├────────────────────────────┤
│"bioprocess_bioprocess" │
├────────────────────────────┤
│"anatomy_anatomy" │
├────────────────────────────┤
│"bioprocess_protein" │
├────────────────────────────┤
│"exposure_disease" │
├────────────────────────────┤
│"exposure_protein" │
├────────────────────────────┤
│"exposure_exposure" │
├────────────────────────────┤
│"exposure_bioprocess" │
├────────────────────────────┤
│"pathway_protein" │
├────────────────────────────┤
│"pathway_pathway" │
├────────────────────────────┤
│"exposure_molfunc" │
├────────────────────────────┤
│"exposure_cellcomp" │
├────────────────────────────┤
│"molfunc_molfunc" │
├────────────────────────────┤
│"cellcomp_cellcomp" │
├────────────────────────────┤
│"anatomy_protein_absent" │
├────────────────────────────┤
│"drug_drug" │
├────────────────────────────┤
│"indication" │
├────────────────────────────┤
│"off-label use" │
├────────────────────────────┤
│"contraindication" │
├────────────────────────────┤
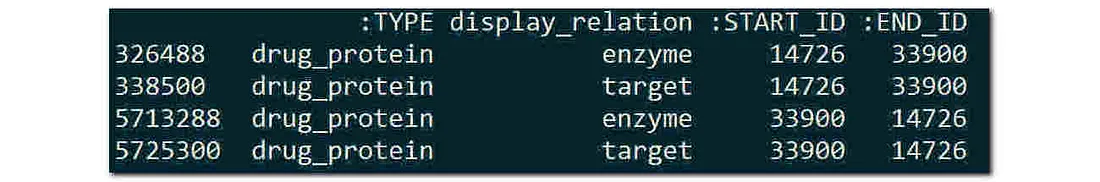
│"drug_protein" │
├────────────────────────────┤
│"disease_phenotype_positive"│
├────────────────────────────┤
│"disease_phenotype_negative"│
├────────────────────────────┤
│"phenotype_phenotype" │
├────────────────────────────┤
│"disease_disease" │
├────────────────────────────┤
│"disease_protein" │
├────────────────────────────┤
│"phenotype_protein" │
└────────────────────────────┘
虽然图表复杂而全面,但我对 "疾病-暴露 "和 "疾病-疾病 "之间的关联很感兴趣。例如,我想知道糖尿病的环境风险因素是什么。查询:
MATCH (e:exposure)-[:exposure_disease]-(d:disease)
WHERE d.node_name CONTAINS 'diabetes'
RETURN d.node_name, e.node_name
ORDER BY d.node_name, e.node_name
结果:
╒═════════════════════════════╤══════════════════════════════════════╕
│d.node_name │e.node_name │
╞═════════════════════════════╪══════════════════════════════════════╡
│"diabetes mellitus (disease)"│"2,4,4',5-tetrachlorobiphenyl" │"diabetes mellitus (disease)"│"2,4,4',5-tetrachlorobiphenyl" │
...
├─────────────────────────────┼──────────────────────────────────────┤
│"diabetes mellitus (disease)"│"bisphenol A" │
├─────────────────────────────┼──────────────────────────────────────┤
│"diabetes mellitus (disease)"│"cyanazine" │
...
对 "双酚 A"(BPA--一种用于生产聚碳酸酯塑料的化学物质)与 "糖尿病 "之间的联系进行一些理智检查:
一些流行病学研究表明,双酚 A 与胰岛素抵抗的发展和葡萄糖稳态受损之间存在显著关联...
备注
原始知识图谱和数据库(PrimeKG由它们组成)肯定会随着时间的推移而改变。
"细节决定成败 合成多个数据源可能会带来挑战,因为不同的数据源可能使用(略有)不同的疾病名称。即使在同一数据源中,"MONDO 包含许多重复的疾病实体,但没有明显的临床相关性。因此,我们将 MONDO 中的疾病归类为医学相关的实体",Chandak 等人[1]指出。然后,作者转而利用 ClinicalBERT 嵌入,以 0.98 的临界值来确定相似性。这只是处理来自不同来源的原始数据的众多步骤之一。
原始的 "edges.csv "包含有向边。起初,我认为这可能是一种假设,因为有向边可以有意义地表示 "A 影响 B"(而不是相反)。然而,当把完整的 edges.csv 加载到 Pandas Dataframe 时,行数完全是论文中预期行数的两倍(8,100,498 对 4,050,249)。因此,我认为作者是碰巧对边缘进行了定向,而不是经过深思熟虑。
总之,PrimeKG 不仅有益于医学研究人员,也有益于机器学习开发人员,因为他们可以利用这种图进行特征工程或图嵌入。