2D数据集视觉指南:解析过采样和欠采样
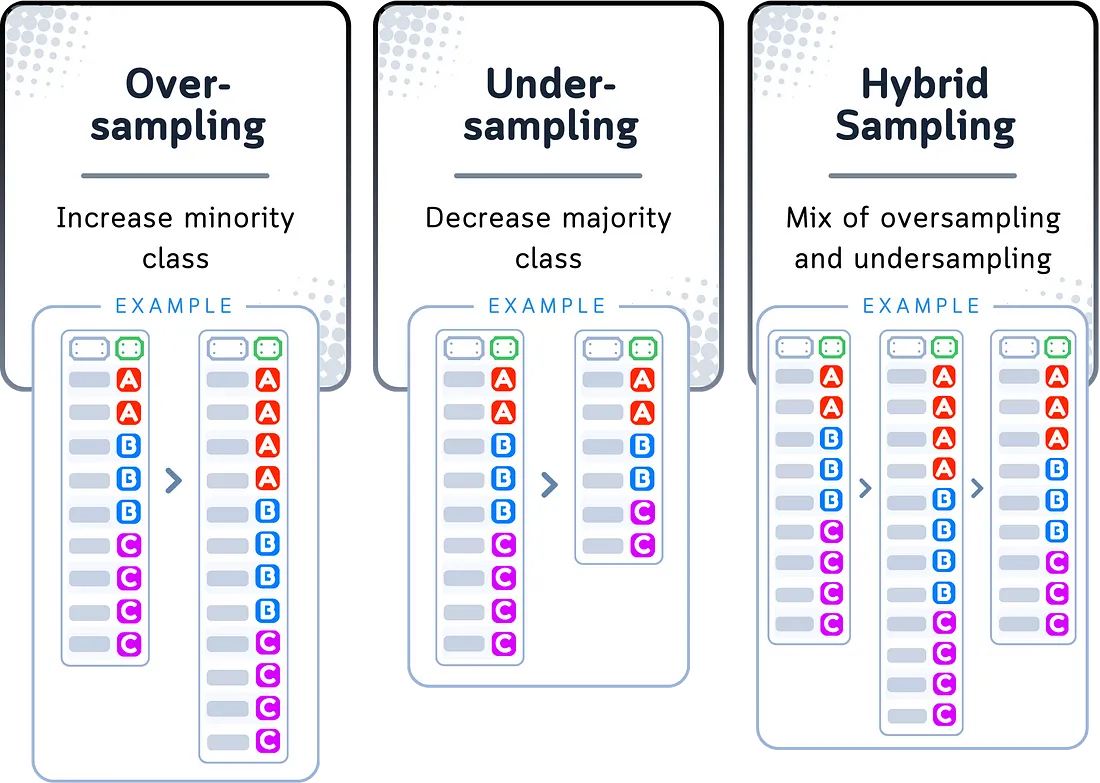
收集每个类别都有完全相同数量的类以进行预测的数据集可能是一个挑战。实际上,事物很少是完全平衡的,当你构建一个分类模型时,这可能会成为一个问题。当模型在这样一个数据集上进行训练时,其中一个类的示例比另一个类多,它通常会变得更擅长预测较大的组,而不太擅长预测较小的组。为了解决这个问题,我们可以使用过采样和欠采样等策略——为较小的组创建更多的示例或从较大的组中删除一些示例。
有许多不同的过采样和欠采样方法(比如具有吓人名称的SMOTE、ADASYN和Tomek Links),但似乎并没有太多资源直观地比较它们的工作原理。因此,在这里,我们将使用一个简单的二维数据集来展示应用这些方法后数据中发生的变化,这样我们就可以看到每种方法的输出有何不同。你将在可视化中看到,这些不同的方法给出了不同的解决方案,谁知道呢,其中一种方法可能适合你的特定机器学习挑战!
定义
过采样
过采样是在一个组的示例远少于另一个组时,使数据集更加平衡的一种方法。它的工作原理是通过复制较小组中示例的更多副本来实现。这有助于数据集更平等地代表两个组。
欠采样
另一方面,欠采样通过从较大的组中删除一些示例,直到其与较小的组大小几乎相同来工作。最终,数据集确实变小了,但两个组的示例数量将更加接近。
混合采样
将过采样和欠采样结合起来可以称为“混合采样”。它通过复制较小组中示例的更多副本来增加该组的大小,同时,也通过删除较大组中一些示例来减少其数量。它试图创建一个更加平衡的数据集——既不太大也不太小。
所用数据集
让我们使用一个简单的人工高尔夫数据集来展示过采样和欠采样的效果。这个数据集展示了在特定的天气条件下,一个人会进行什么样的高尔夫活动。
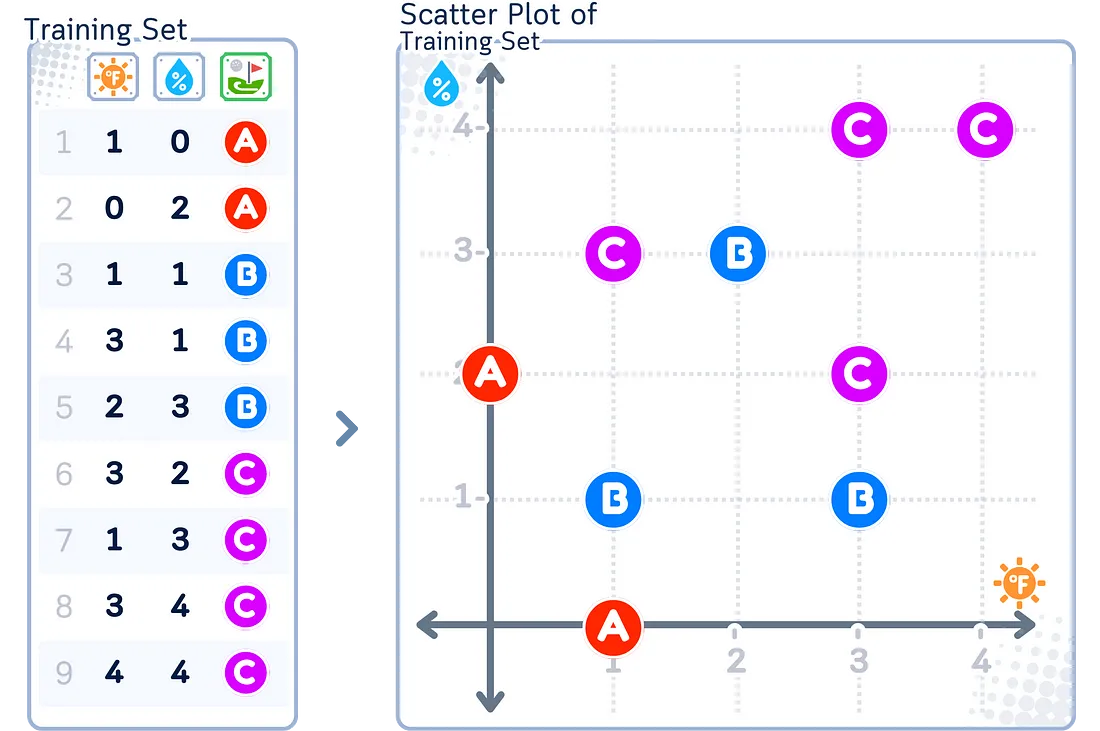
请注意,虽然这个小数据集有助于理解这些概念,但在实际应用中,在应用这些技术之前,你需要更大的数据集,因为样本过少可能会导致结果不可靠。
过采样方法
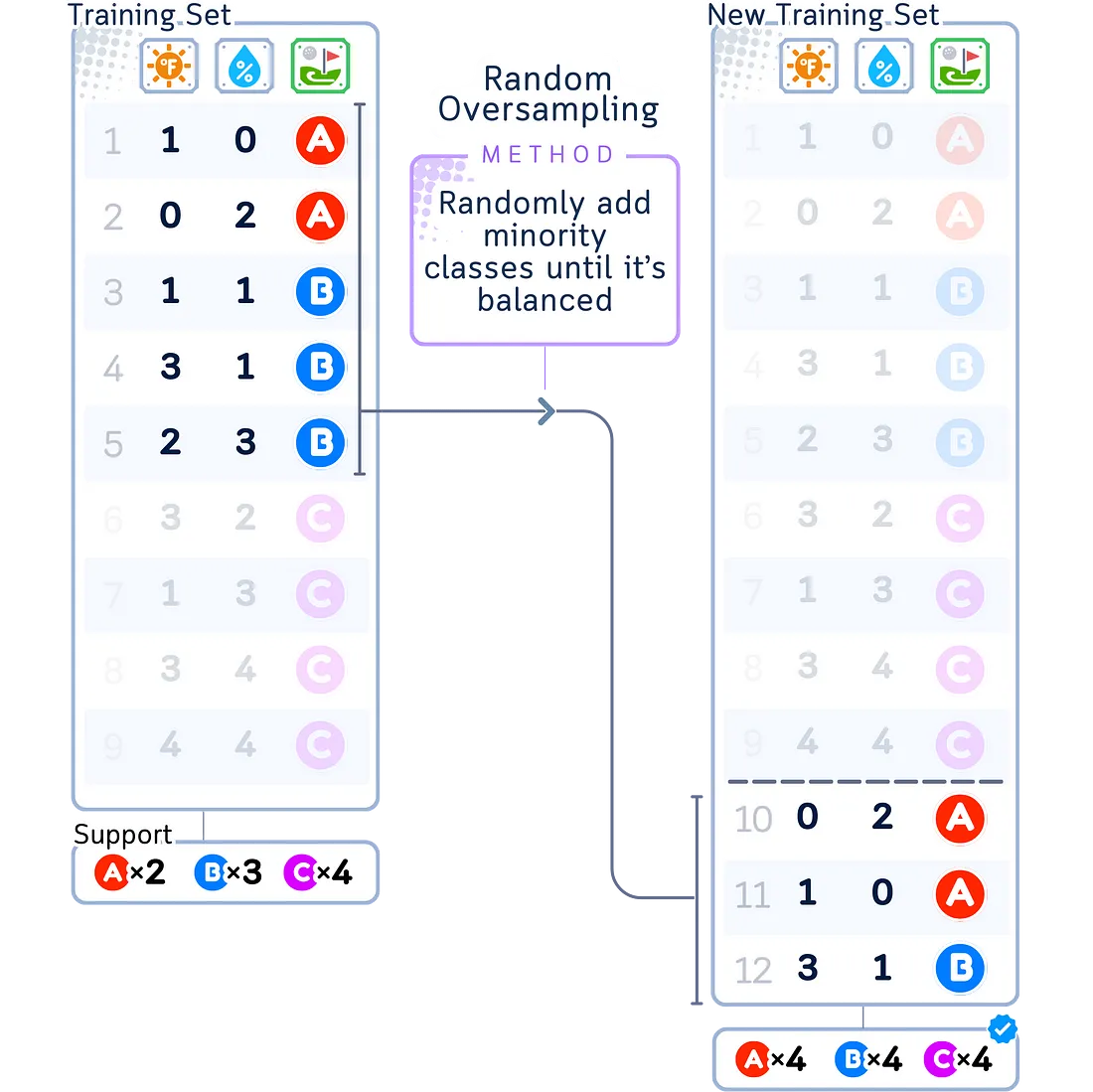
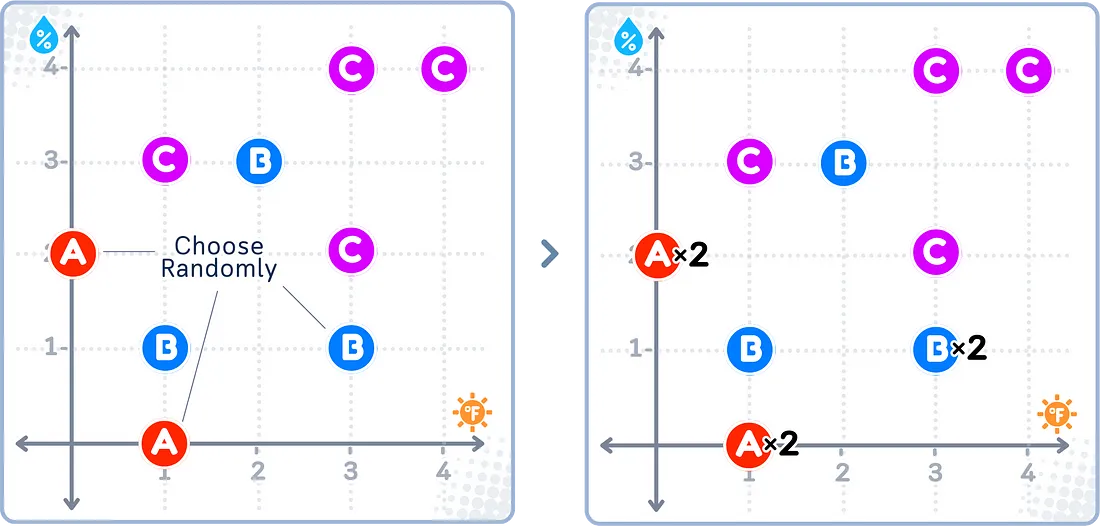
随机过采样
随机过采样是一种简单的使较小组变大的方法。它的工作原理是复制较小组中的示例,直到所有类别都平衡为止。
- 最适合需要快速平衡的非常小的数据集
- 不推荐用于复杂的数据集
SMOTE
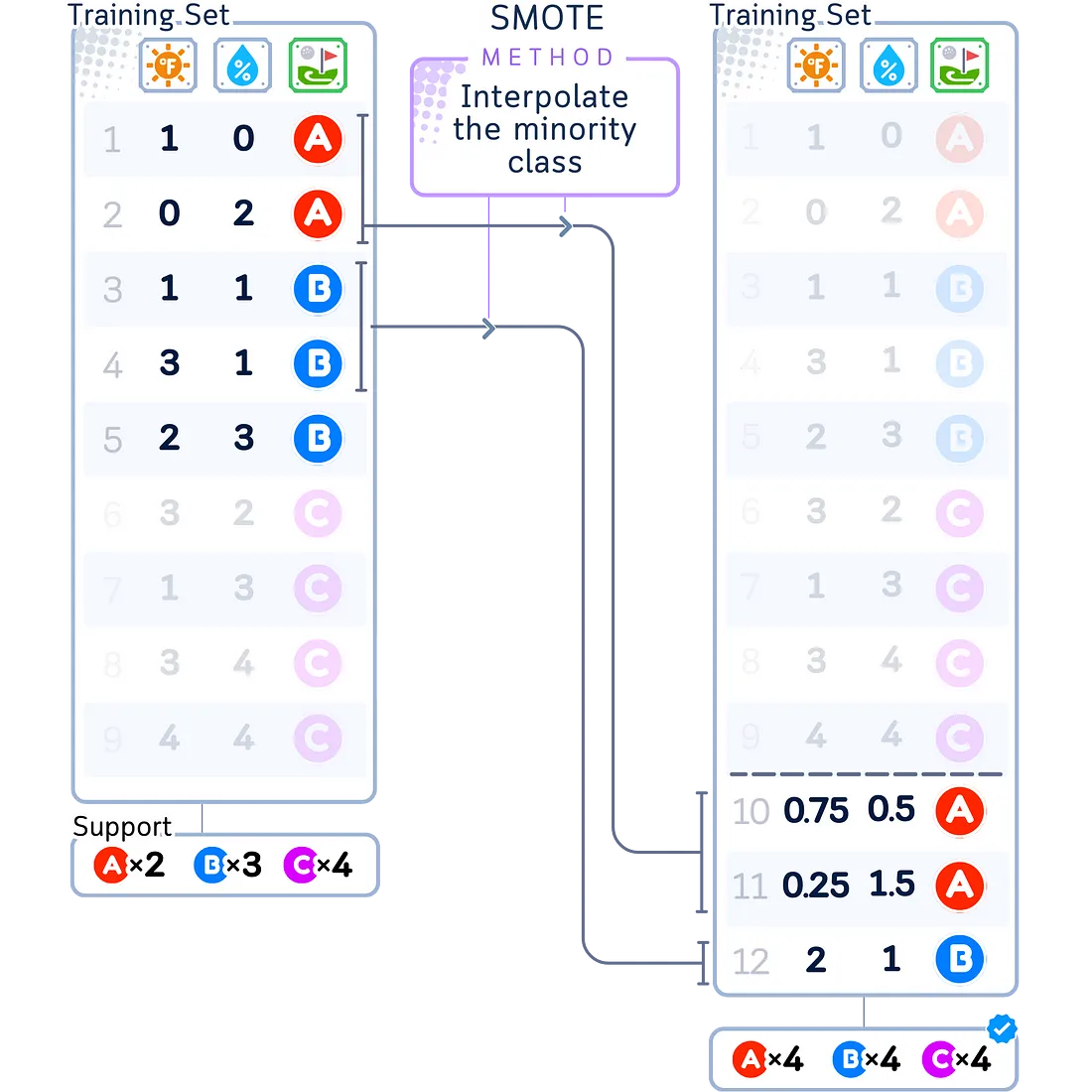
SMOTE(合成少数类过采样技术)是一种过采样技术,它通过插值较小组的样本来生成新的样本。与随机过采样不同,它不仅仅复制现有的样本,而是利用较小组中的样本来生成它们之间的一些新样本。
- 当你拥有相当数量的样本可供使用并且需要数据具有多样性时效果最佳
- 如果你只有非常少的样本,则不推荐
- 如果数据点过于分散或嘈杂,则不推荐

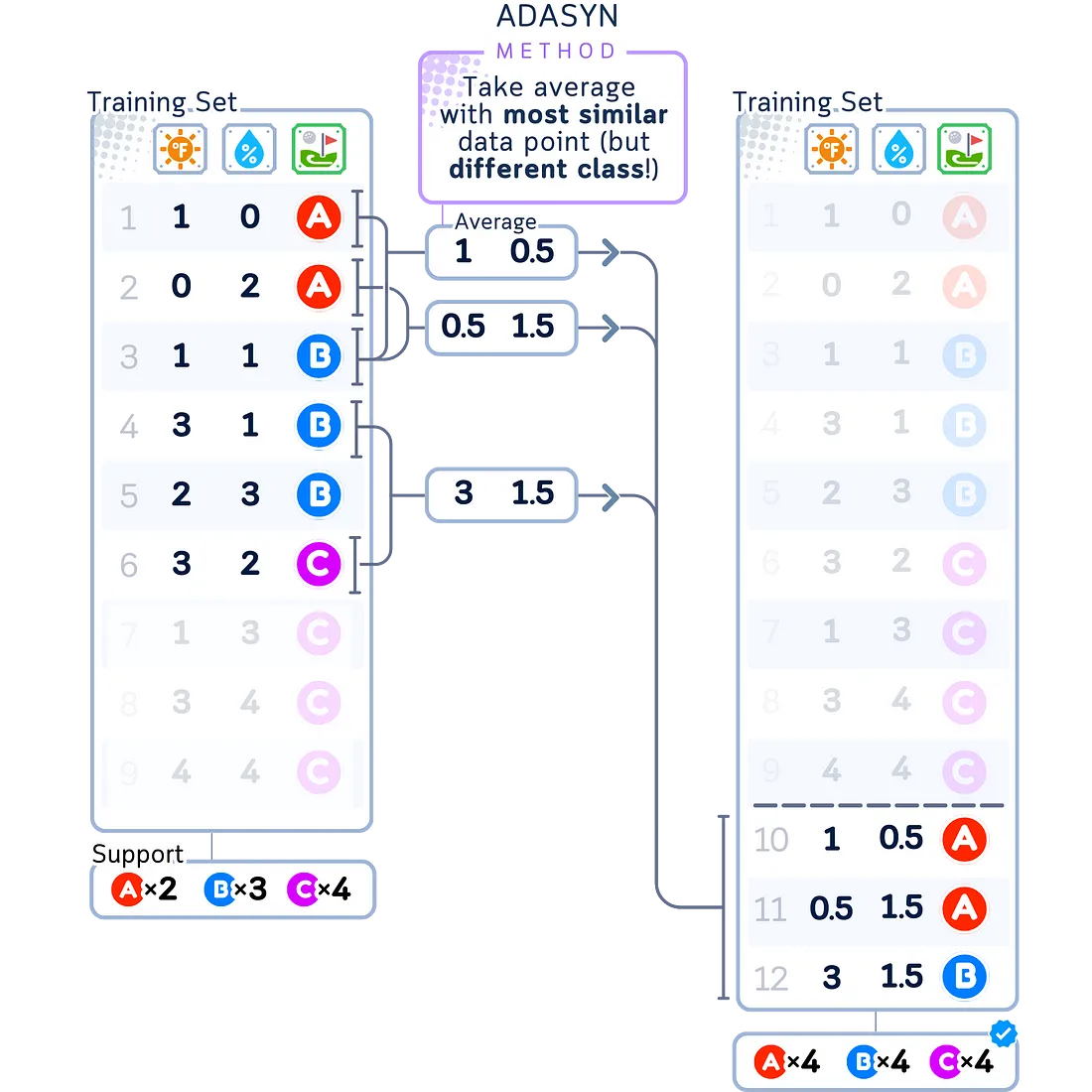
ADASYN
ADASYN(自适应合成)类似于SMOTE,但它更侧重于在较小组中较难学习的部分生成新样本。它会找到最难分类的样本,并在这些样本周围生成更多的新点。这有助于模型更好地理解具有挑战性的区域。
- 当你的数据中某些部分比其他部分更难分类时效果最佳
- 对于具有挑战性区域的复杂数据集效果最佳
- 如果你的数据相对简单且直接,则不推荐
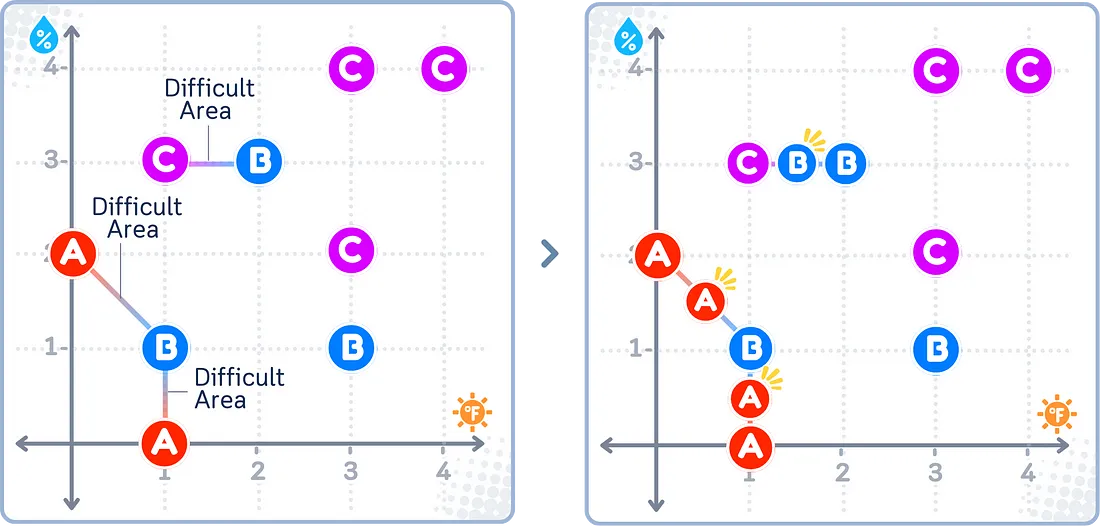
欠采样方法
欠采样通过缩小较大组的规模,使其大小更接近较小组。以下是实现这一目标的一些方法:
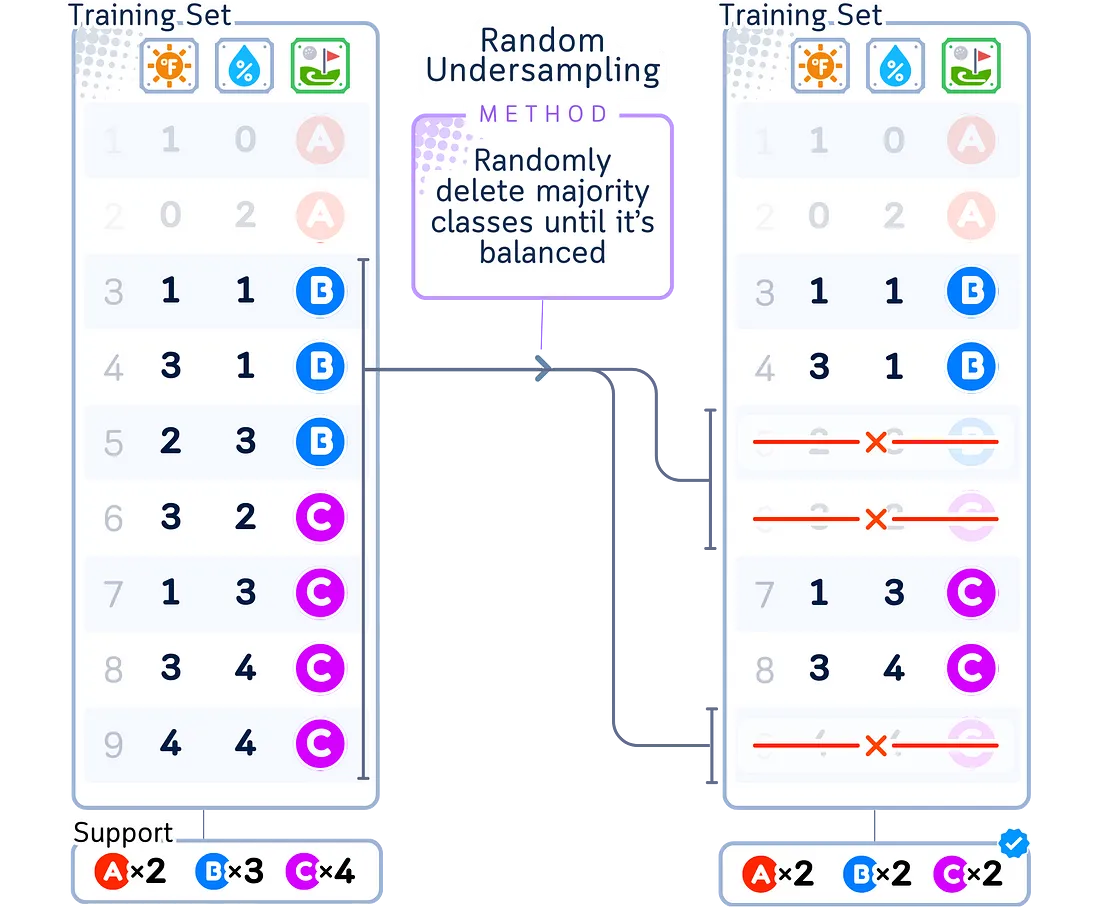
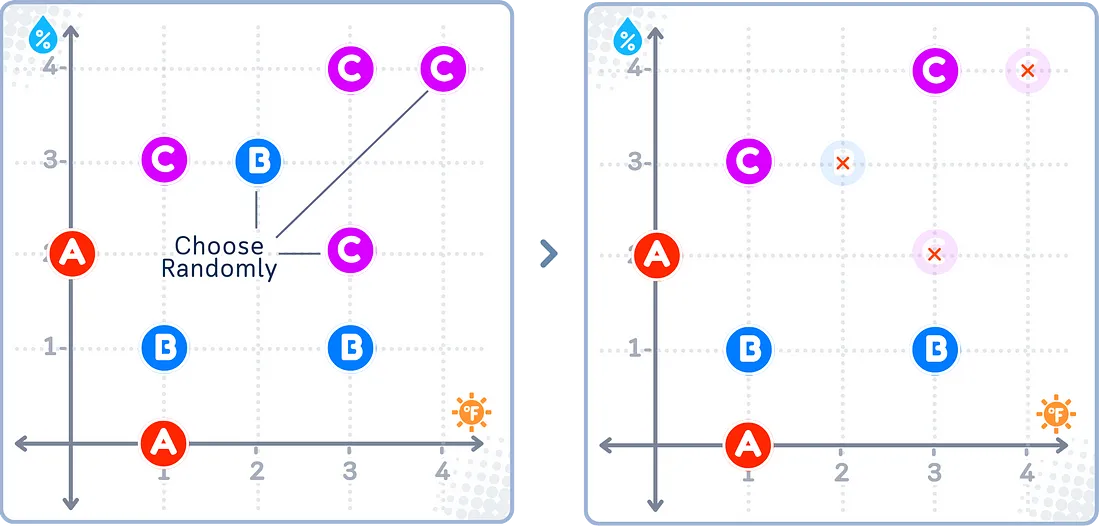
随机欠采样
随机欠采样会随机删除较大组中的样本,直到其与较小组大小相同。就像随机过采样一样,这种方法非常简单,但可能会消除真正显示组别差异的重要信息。
- 最适合包含大量重复样本的非常大数据集
- 当你需要一个快速、简单的解决方案时效果最佳
- 如果你的较大组中的每个样本都很重要,则不推荐
- 如果你不能承受任何信息的丢失,则不推荐
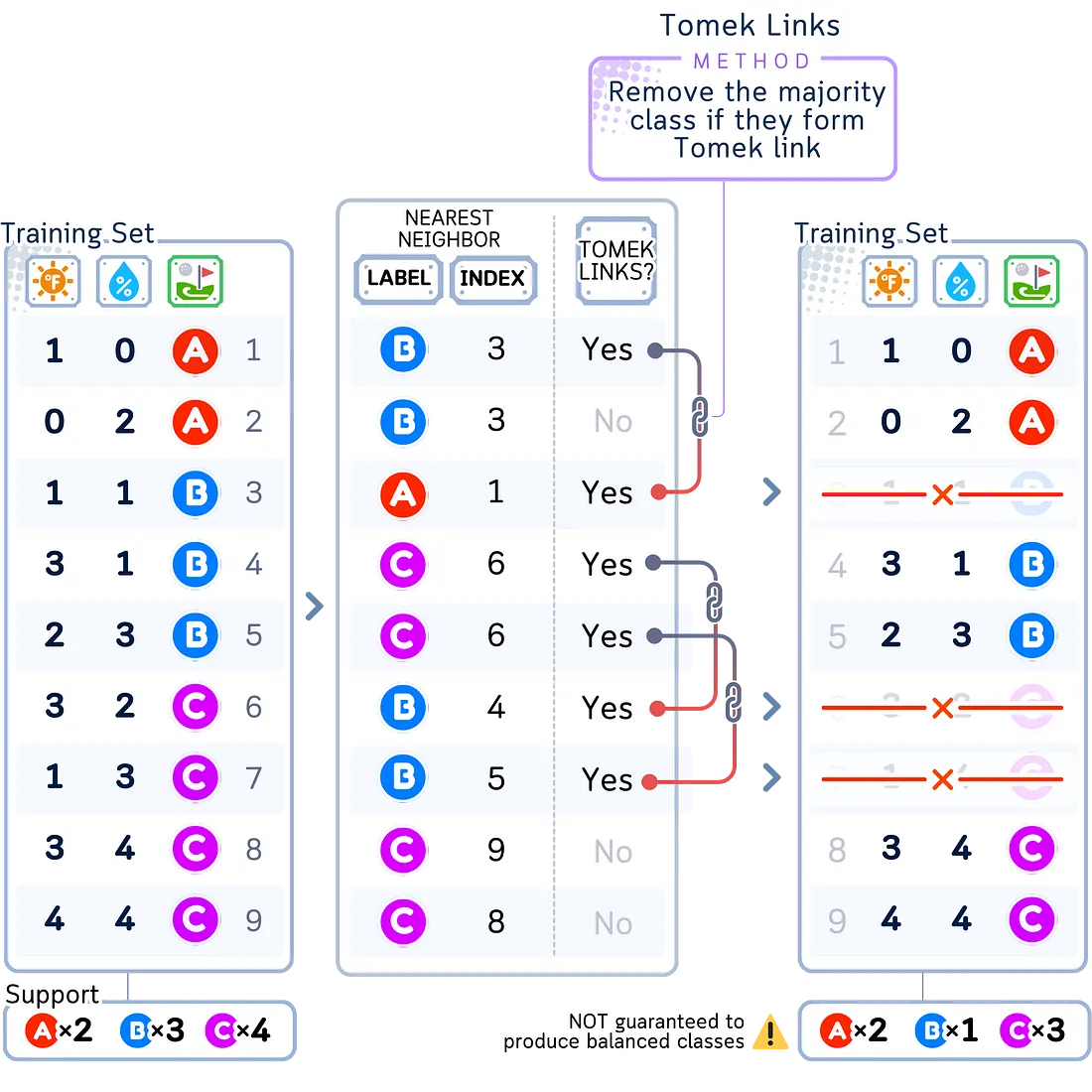
Tomek链接
Tomek链接是一种欠采样方法,它可以使组之间的“界限”更加清晰。它搜索来自不同组但非常相似的样本对。当找到一个样本对,其中两个样本是彼此的最近邻但属于不同组时,它会删除较大组中的那个样本。
- 当你的组别重叠过多时效果最佳
- 对于清理混乱或嘈杂的数据效果最佳
- 当你需要组别之间有清晰的界限时效果最佳
- 如果你的组别已经分离得很好,则不推荐

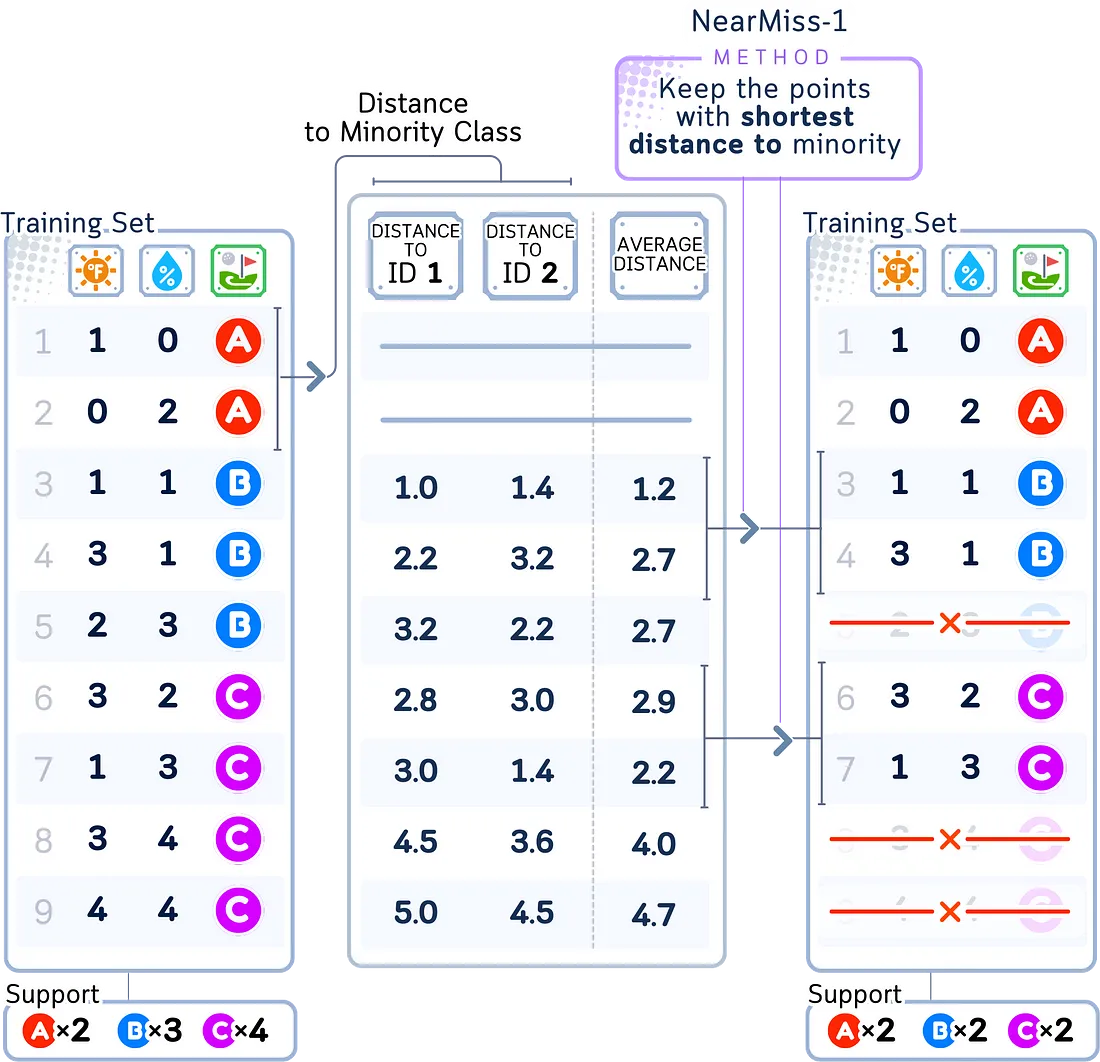
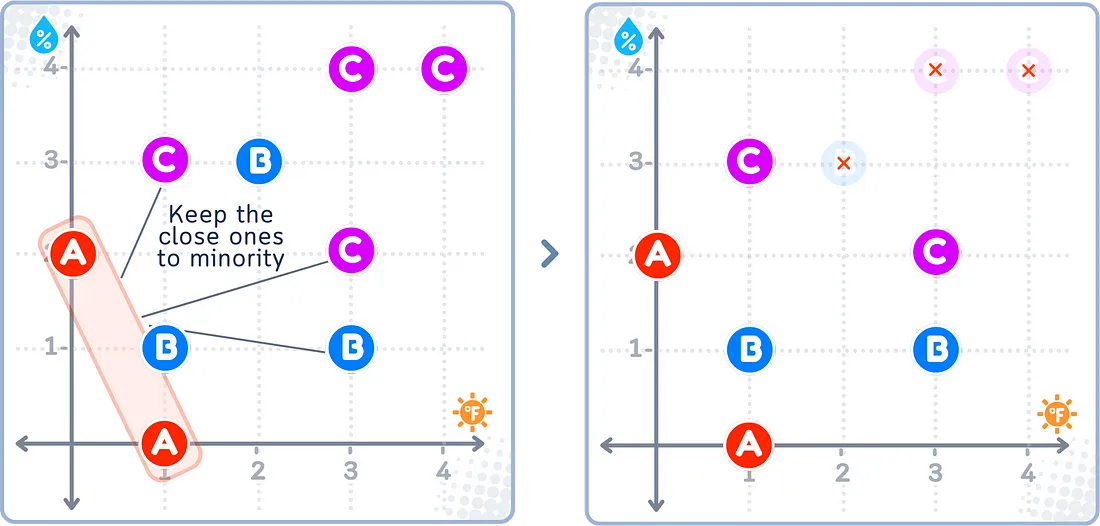
Near Miss
Near Miss 是一系列基于不同规则的欠采样技术:
- Near Miss-1:保留较大组中距离较小组中样本最近的样本。
- Near Miss-2:保留较大组中与其在较小组中三个最近邻的平均距离最小的样本。
- Near Miss-3:保留较大组中距离其所在组内其他样本最远的样本。
这里的主要思路是保留较大组中最具信息量的样本,并删除那些不太重要的样本。
- 当你想要控制保留哪些样本时效果最佳
- 如果你需要一个简单、快速的解决方案,则不推荐
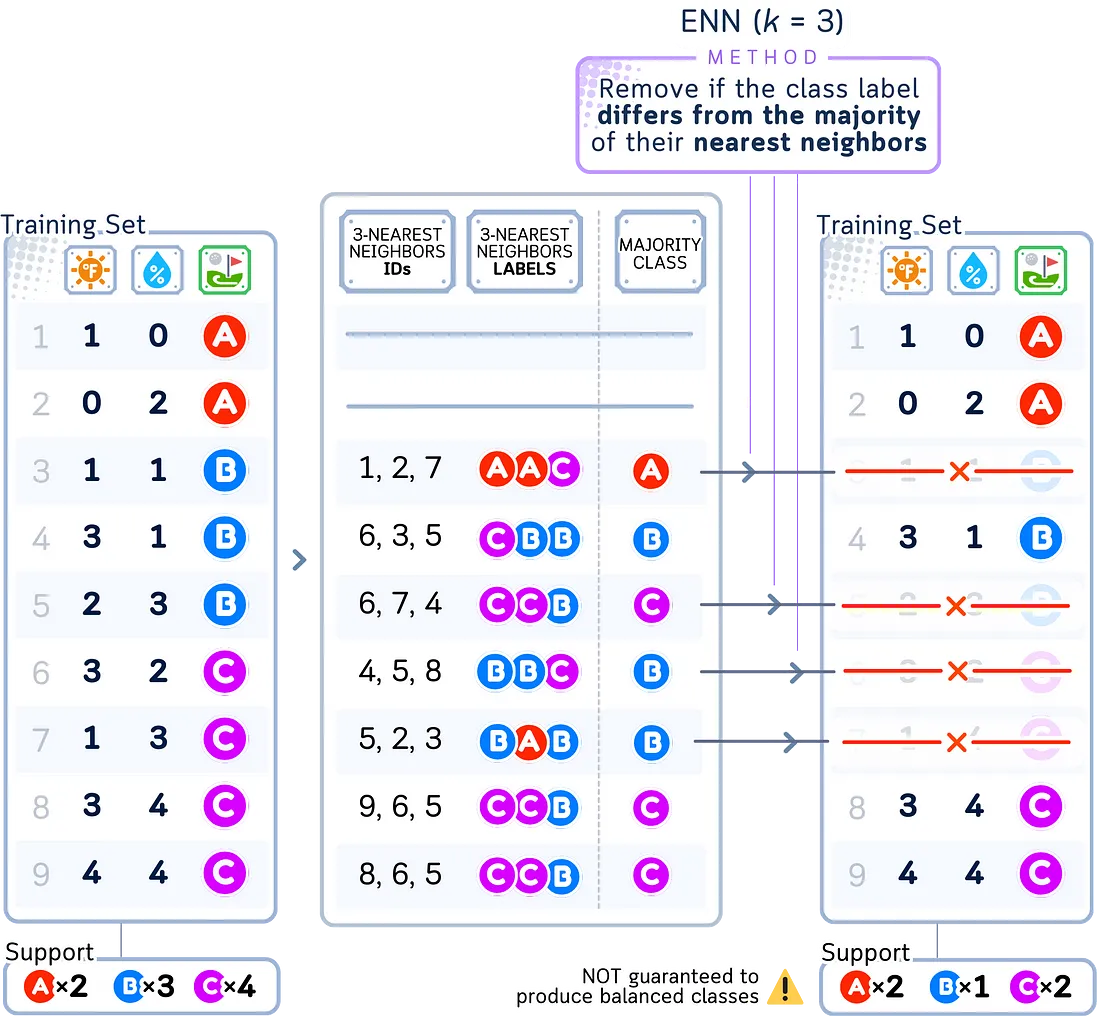
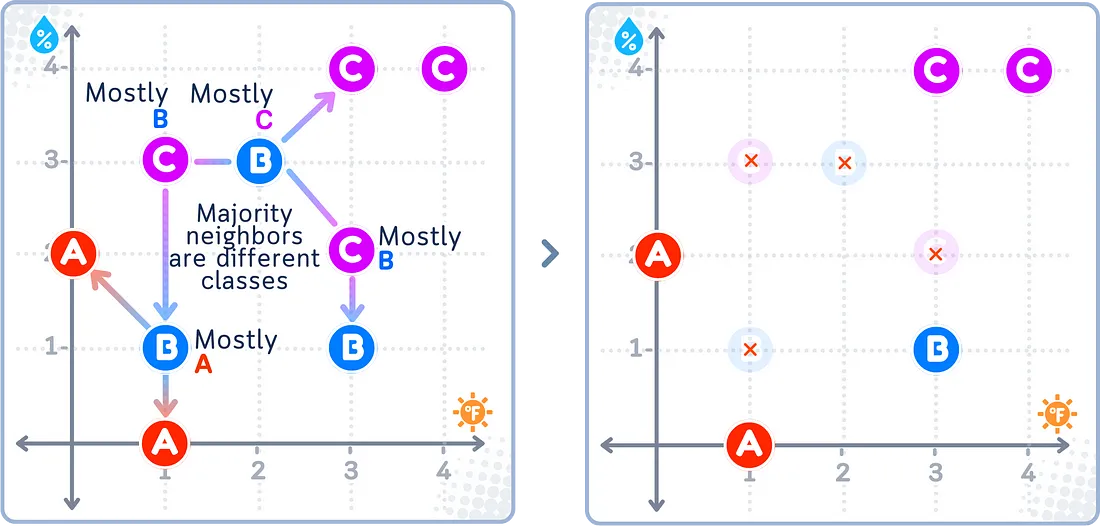
ENN
编辑最近邻(ENN)方法会删除可能是噪声或离群点的样本。对于较大组中的每个样本,它会检查其大多数最近邻是否属于同一组。如果不是,则删除该样本。这有助于在组之间创建更清晰的界限。
- 最适合清理混乱数据
- 当你需要移除离群点时效果最佳
- 最适合创建更清晰的组界限
- 如果你的数据已经干净且组织良好,则不推荐
混合采样方法
SMOTETomek
SMOTETomek首先使用SMOTE为较小组创建新样本,然后使用Tomek链接删除“令人困惑”的样本来清理混乱的边界。这有助于创建一个更平衡的数据集,具有更清晰的边界和更少的噪声。
- 最适合极度不平衡的数据
- 当你既需要更多样本又需要更清晰的边界时效果最佳
- 在处理嘈杂、重叠的组时效果最佳
- 如果你的数据已经干净且组织良好,则不推荐
- 不推荐用于小数据集
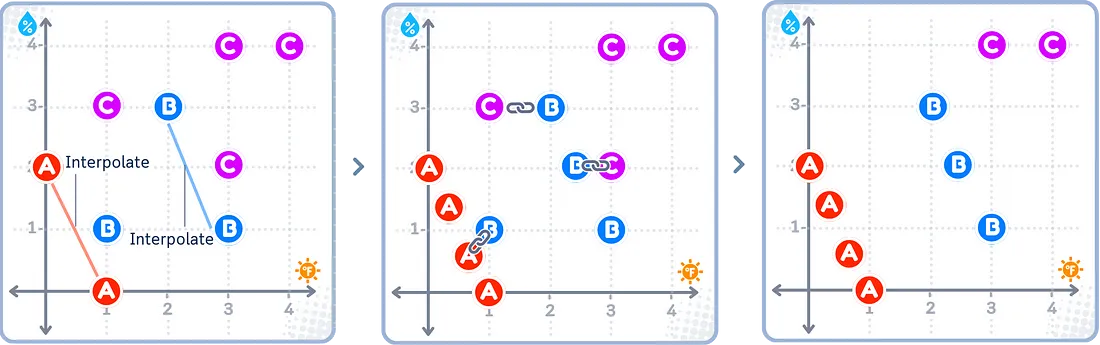
SMOTEENN
SMOTEENN首先使用SMOTE为较小组创建新样本,然后使用ENN删除与邻居不匹配的样本,从而清理两个组。与SMOTETomek类似,这有助于创建一个更干净的数据集,并使组之间的边界更清晰。
- 最适合同时清理两个组
- 当你需要更多样本但数据需要更干净时效果最佳
- 在处理大量离群点时效果最佳
- 如果你的数据已经干净且组织良好,则不推荐
- 对于小数据集不推荐
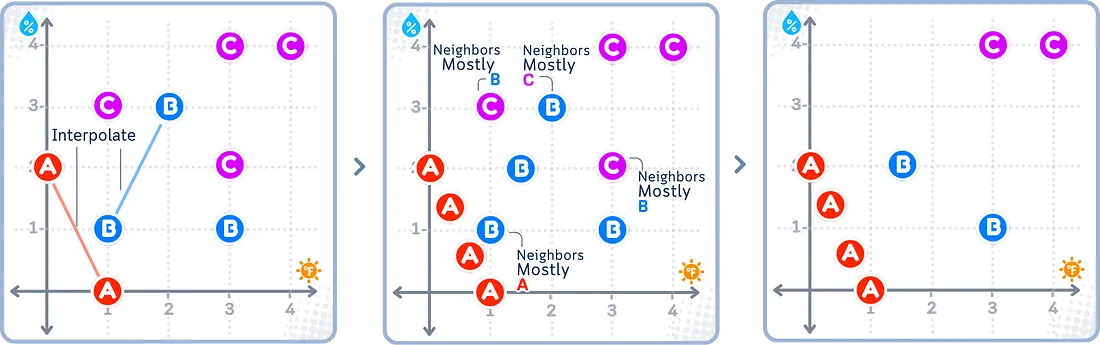
使用重采样方法的风险
重采样方法可能有所帮助,但也存在一些潜在风险:
过采样:
- 制作人工样本可能会产生现实生活中不存在的错误模式。
- 由于合成样本的存在,模型可能会变得过于自信。这将导致在实际应用时出现严重失败。
- 如果重采样操作不当(例如在交叉验证前分割数据之前进行重采样),则存在数据泄漏的风险。
欠采样:
- 你可能会永久丢失重要信息。
- 你可能会意外破坏类别之间的重要边界,从而导致对问题的误解。
- 你可能会创建出与现实世界条件相比差异过大的人工类别分布。
混合方法:
- 结合两种方法的错误可能会使情况变得更糟,而不是更好。
在使用重采样方法时,很难在获取类别不平衡的同时不改变数据中重要模式之间找到正确的平衡。根据我的经验,不正确的重采样实际上可能会损害模型性能,而不是提高它。
在转向重采样之前,请尝试使用自然能更好处理不平衡数据的模型,如基于树的算法。重采样应该是更广泛策略的一部分,而不是解决类别不平衡问题的唯一解决方案。
过采样与欠采样代码总结
对于代码示例,我们将使用imblearn库提供的方法:
import pandas as pd
from imblearn.over_sampling import SMOTE, ADASYN, RandomOverSampler
from imblearn.under_sampling import TomekLinks, NearMiss, RandomUnderSampler
from imblearn.combine import SMOTETomek, SMOTEENN
# Create a DataFrame from the dataset
data = {
'Temperature': [1, 0, 1, 3, 2, 3, 1, 3, 4],
'Humidity': [0, 2, 1, 1, 3, 2, 3, 4, 4],
'Activity': ['A', 'A', 'B', 'B', 'B', 'C', 'C', 'C', 'C']
}
df = pd.DataFrame(data)
# Split the data into features (X) and target (y)
X, y = df[['Temperature', 'Humidity']], df['Activity'].astype('category')
# Initialize a resampling method
# sampler = RandomOverSampler() # Random OverSampler for oversampling
sampler = SMOTE() # SMOTE for oversampling
# sampler = ADASYN() # ADASYN for oversampling
# sampler = RandomUnderSampler() # Random UnderSampler for undersampling
# sampler = TomekLinks() # Tomek Links for undersampling
# sampler = NearMiss(version=1) # NearMiss-1 for undersampling
# sampler = EditedNearestNeighbours() # ENN for undersampling
# sampler = SMOTETomek() # SMOTETomek for a combination of oversampling & undersampling
# sampler = SMOTEENN() # SMOTEENN for a combination of oversampling & undersampling
# Apply the resampling method
X_resampled, y_resampled = sampler.fit_resample(X, y)
# Print the resampled dataset
print("Resampled dataset:")
print(X_resampled)
print(y_resampled)