使用LLM创建ICD代码知识图谱
前往医院咨询医生和医疗从业者自身健康问题的患者,其诊断、治疗过程和其他医疗细节会被记录为电子健康记录(EHR)。以美国为例,办公室医生使用EHR的比例已从2008年的42%飙升至2021年的超过88%,导致现在大部分医疗信息都以这种格式存储。这些EHR被用于各种下游活动,如ICD编码,即将医疗诊断和状况与标准化、唯一的字母数字代码相关联。
鉴于EHR以文本形式数字化组织,我们可以使用自动化方法来识别文本中的医疗信息和实体,并尝试将它们映射到相应的ICD代码。然而,ICD编码是一个复杂的过程——它需要准确识别EHR中的诊断,将其与其他相关的医疗信息和实体相关联,然后映射到正确的ICD代码。这项任务需要强大的自然语言理解(NLU)能力以及对ICD分类法的详细了解。通常,这些任务由具有分配ICD代码所需专业知识的医疗从业者和临床编码员执行。在这篇文章中,我们探讨了如何将ICD代码表示为知识图谱(KG),以及如何构建这个KG。
我们能否直接应用大型语言模型(LLM)进行ICD编码?
大型语言模型越来越多地用于各种临床和医疗应用,如医疗报告摘要和实体提取。这是基于近期研究表明,大型语言模型在医学资格考试中表现出色,并且在医学问答任务上与医疗从业者和医生相当,甚至优于他们。
然而,ICD编码与摘要或实体提取等任务有显著不同。如前所述,ICD编码需要强大的自然语言理解能力,以及对每个代码所代表内容的深入理解。在ICD编码的上下文中,自然语言理解涉及实体提取和关系提取等任务,以识别文本中医疗实体之间的关系。
大型语言模型在零样本设置下的实体提取和关系提取方面表现良好。然而,它们不能完全依赖其内部化的参数化知识来选择正确的ICD代码。ICD分类法包含超过70,000个层次结构化的代码,这意味着大型语言模型必须在这个庞大且有序的输出空间中精确映射临床诊断——这是一项极具挑战性的任务。
在实践中,鉴于ICD代码的数量庞大,当仅凭其参数化知识要求大型语言模型预测ICD代码时,它们很可能会产生幻觉。我们通过向GPT-4o提供ICD代码描述并要求其预测相应的ICD代码,并观察到幻觉的实例来证明这一点。在最左边的例子中,我们用ICD代码S46.301的描述“右臂三头肌、筋膜和肌腱的不明损伤”向大型语言模型提供提示。在右边的例子中,我们使用了ICD代码S48.11的描述“右肩和肘部之间水平的完全创伤性截肢”。

完全依赖大型语言模型(LLM)以全自动、端到端的方式进行ICD编码可能会带来挑战。
我们通过将ICD编码分解为语言解析和外部知识查询来处理它。特别地,我们探索了如何将ICD代码表示为知识图谱(Knowledge Graph),该图谱稍后可以与LLM集成,作为在检索增强生成(RAG)设置中查询ICD代码的外部知识。此外,我们还展示了如何利用LLM在命名实体识别(NER)和关系提取等任务中的零样本能力来高效地构建这个知识图谱。
什么是知识图谱?
图到底是什么?简单来说,图是一种数据结构,它帮助我们映射出不同实体(无论是人、设备还是医疗代码)之间的连接。从结构上看,图由节点和边组成。节点代表实体本身,而边则表示它们之间的关系或交互。
图使得复杂系统(如社交网络,其中每个节点可能代表一个人,每条边代表一段友谊;或计算机网络,其中节点是设备,边是连接它们的链路)的可视化和分析变得容易。总的来说,它们是表示连接的数据以及不同实体之间关系的强大方法。
然而,简单的图并不提供关于实体之间可能存在的特定类型和性质的关系的任何信息。例如,给定代表A和B两个人的两个实体,一个基本的图可能只是表明这两个人是连接的,而不揭示他们连接的性质。这是一种家庭关系、职业关系还是友谊?
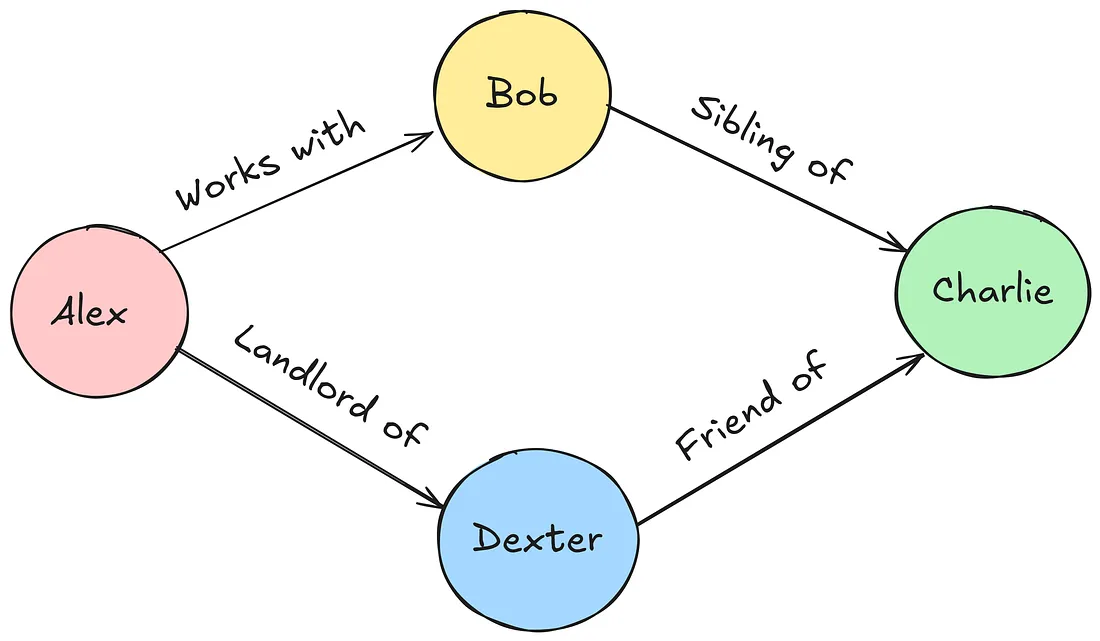
这就是知识图谱(Knowledge Graph,简称KG)发挥作用的地方。知识图谱通过嵌入语义信息——定义节点之间关系意义的额外细节——来增强基本的图结构。与简单的图不同,知识图谱能够捕捉层次结构、属性以及实体之间更复杂、更微妙的关系。
为何使用知识图谱进行ICD编码?
为何使用知识图谱进行ICD编码?ICD编码提供了一个详细的框架,用于以层次结构的方式对疾病、症状、状况和医疗程序进行分类,其中广泛的类别会细分为越来越具体的子类型。知识图谱非常适合这种结构,因为它们能够建模这些复杂的关系。知识图谱可以自然地捕捉父子结构,并反映不同代码之间的关系。这使得它们成为表示ICD分类体系的理想选择。
除了简单捕捉这些层次关系外,知识图谱还通过链接ICD代码所表示的相互关联的实体提供了另一个好处。例如,知识图谱不仅可以描绘ICD代码的结构,还可以描绘各种疾病、症状和程序之间的关系。这使我们能够更好地理解特定疾病与其相关症状、治疗方法或风险因素之间的关系。
知识图谱可以与大型语言模型(LLM)集成,特别是在检索增强生成(RAG)和访问外部知识源的上下文中。RAG将LLM与外部知识库相结合,以提高响应的准确性和相关性。在这种设置中,知识图谱作为LLM在生成过程中可以查询的结构化知识库,提供更具上下文感知和事实基础的输出。
创建知识图谱
我们如何为ICD代码创建知识图谱?每个ICD代码通常都由一个捕获代码本质的纯文本描述来表示。使用这些描述,我们为每个ICD代码构建一个图,以捕捉其关键关系和属性。一旦创建了所有单个ICD代码图,我们将它们合并成一个表示整个ICD编码系统的统一知识图谱。
图创建过程
知识图谱的构建涉及三个主要步骤:
- 命名实体识别(NER)
- 关系抽取(RE)
- 实体链接(EL)
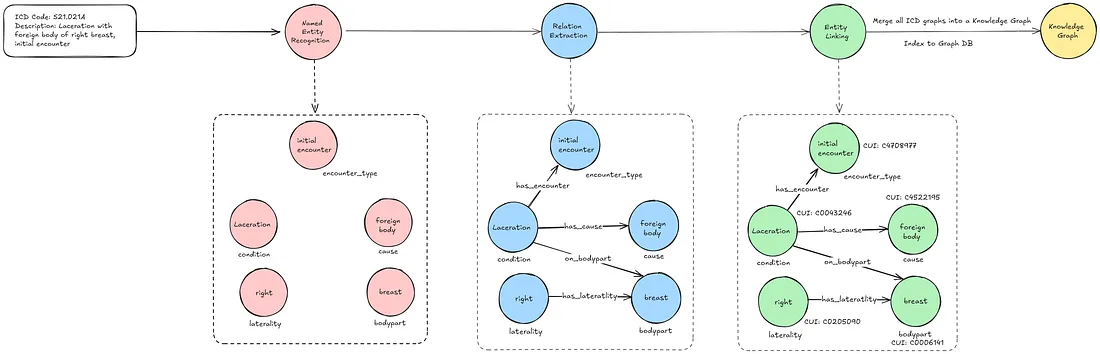
命名实体识别
命名实体识别(NER)是从非结构化文本中识别和提取实体的过程。在ICD编码的上下文中,NER帮助我们在临床文档中找到并分类关键的医学术语和实体,如疾病、症状和治疗方法。例如,NER可以识别“糖尿病”为一种疾病,而“胸痛”为一种症状。
NER是知识图谱构建过程中的重要步骤,因为它帮助我们确定最终图中的节点。我们用于NER的工具必须高度准确,因为错误的检测和误差会给我们的知识图谱增加噪声。
关系抽取
在识别出实体后,下一步是关系抽取——确定不同实体之间是如何相互关联的。在ICD编码的上下文中,这可能包括医学实体之间的链接,如症状和状况,或疾病和治疗。例如,可能存在“胸痛”这一状况与可能的诊断“心肌梗死”之间的关系。这一步对于在图中构建我们的节点(实体)之间的边(关系)至关重要。
实体链接
实体链接通过将识别出的实体和关系与特定概念相连接,确保它们清晰且一致。在医学背景下,许多术语有同义词或变体,如“心脏病发作”和“心肌梗死”。实体链接将这些术语对齐到一个统一的概念上。这一步有助于保持图的一致性,并允许进行更准确的查询。
图的存储和查询
一旦图创建完成,就需要将其存储在一个能够高效处理图数据并允许轻松查询的数据库中。我们可以使用图数据库,它设计用于存储节点和边(代表实体及其关系)来存储我们的图。这使我们能够从图中查询和检索信息。
构建ICD图
如何利用LLM创建知识图谱?
我们在图创建的初始阶段使用LLM来识别命名实体和将它们联系起来的关系。对于实体检测,我们观察到像Scispacy这样的库中的模型并不完全覆盖我们构建图所需提取的实体类型。Scispacy的默认实体提取模型可以从文本中识别所有医学实体,但不提供实体类型。我们使用LLM来完成这些任务,因为它们允许我们灵活地描述我们想要提取和链接的特定实体,而不需要专门的训练。
命名实体识别
第一步是确定需要提取哪些特定类型的实体。医疗状况、身体部位和症状是显而易见的候选类别,但考虑额外的实体类型也很重要。
为此,我们首先随机抽取一大批ICD描述,并提示LLM建议一套全面的相关实体类型以供提取。基于这种分析,我们确定了12个主要实体类型,包括:
- 状况
- 身体部位
- 严重程度
- 接诊类型
- 原因
- 侧别
- 人员
- 胎儿
- 手术/程序
- 并发症
- 孕期(三个月为一期)
- 其他信息
这些实体类型代表了可以在ICD代码描述中找到的医学实体的范围。在我们用于执行NER和RE的提示中,可以找到对每个实体类型所代表内容的解释。
关系抽取
在从ICD描述中识别出实体后,我们继续识别和链接相关实体。为此,我们考虑医学实体之间的所有可能关系。然而,在大多数情况下,主要锚点是对应于ICD代码的诊断,它作为其他实体围绕其链接的中心实体。
为NER和关系抽取构建提示
我们使用LLM(利用GPT-4o mini LLM)在一次调用中对ICD描述执行命名实体识别(NER)和关系抽取(RE)。第一步是构建一个有效的提示。
构建提示需要:
- 定义实体:我们需要明确指定我们想要提取的实体类型,并为每个类别提供精确的描述。
- 提供示例:我们需要包含代表性的ICD描述,并使用少样本方法说明输出应该如何看起来。
- 结构化格式:我们必须为LLM定义一个结构化的输出格式,以便我们能够正确解析必要的信息。
我们首先概述实体提取任务,指定实体类型并附带示例。接下来,我们通过描述实体之间的潜在关系并提供相关示例来定义关系抽取任务。最后,我们包括四个少样本示例来演示所需的输出格式。
prompt_relation_extraction = """You are an expert medical professional, qualified in ICD coding and medical terminology."""You are an expert medical professional, qualified in ICD coding and medical terminology.
You are given a description of an ICD code, and your task is to extract relevant entities and construct a graph by identifying the relationships between these entities.
Follow these detailed instructions:
Step 1: Extract Entities
Identify and extract the following entity types from the provided ICD code description.
Instructions:
1. Do not output any placeholders for entity types that are not mentioned in the description.
2. You must separate entities of the same type with ||.
3. Strictly do not output content that is not present in the description.
Entity Types:
1. condition: Identify the medical conditions and/or injuries described by the ICD code.
2. bodypart: Identify any specific body parts mentioned affected by the conditions.
3. severity: Determine the severity or degree or stage of the conditions if mentioned (e.g., first degree, second degree, third degree, mild, moderate, severe, type I, type II).
4. encounter_type: Identify the type of medical encounter described (e.g., initial encounter, subsequent encounter, sequela).
5. cause: Extract the cause or reasons for the conditions or injuries (e.g., foreign object, poisoning, burn, fall, collision).
6. laterality: Identify if a specific side of the body is affected (e.g., left, right, unspecified).
7. person: Represents the person affected (e.g., unspecified person, suspect, bystander).
8. fetus: Represents the fetus affected in maternal care cases.
9. procedure: Represents any treatment or procedures associated with the conditions (e.g., surgery, prosthesis).
10. complication: Represents any complications arising from the conditions or treatments (e.g., nonunion, delayed healing).
11. trimester: Represents the trimester of pregnancy if applicable.
12. other_info: Represents any other important medical information that are not covered by the above entities.
Step 2: Construct Relationships
From the extracted entities, identify and extract the entities that are related to each other.
Some examples of relations are:
- Example: "Dislocation" (condition) affects "knee" (bodypart).
- Example: "Burn" (condition) has "third degree" (severity).
- Example: "Pre-eclampsia" (condition) occurs during "second trimester" (trimester).
- Example: "Laceration" (condition) caused by "sharp object" (cause).
- Example: "hand" (bodypart) affected is "right" (laterality).
- Example: "Injury" (condition) affects "unspecified person" (person).
- Example: "Chorioamnionitis" (condition) affects "fetus" (fetus).
- Example: "Gastric band complication" (condition) treated by "gastric band procedure" (procedure).
- Example: "Dislocation" (condition) occurs during "initial encounter" (encounter_type).
- Example: "Burn" (condition) has "delayed healing" (complication).
Represent each pair of related entities in the format (Entity 1||Entity 2).
Some examples of input and output are provided below:
### Input
ICD Code Description: Laceration with foreign body of right breast, initial encounter
### Output
Entities:
condition: Laceration
bodypart: breast
encounter_type: initial encounter
cause: foreign body
laterality: right
Relations:
(Laceration||foreign body)
(Laceration||breast)
(right||breast)
(Laceration||initial encounter)
### Input
ICD Code Description: Nondisplaced segmental fracture of shaft of radius, unspecified arm, subsequent encounter for open fracture type I or II with delayed healing
### Output
Entities:
condition: Nondisplaced segmental fracture||open fracture
bodypart: shaft of radius||arm
encounter_type: subsequent encounter
severity: type I||type II
complication: delayed healing
laterality: unspecified
Relations:
(Nondisplaced segmental fracture||shaft of radius)
(Nondisplaced segmental fracture||arm)
(unspecified||arm)
(Nondisplaced segmental fracture||subsequent encounter)
(subsequent encounter||open fracture)
(open fracture||type I)
(open fracture||type II)
(open fracture||delayed healing)
### Input
ICD Code Description: Displaced oblique fracture of shaft of left fibula, subsequent encounter for open fracture type IIIA, IIIB, or IIIC with malunion
### Output
Entities:
condition: Displaced oblique fracture||open fracture
bodypart: shaft||fibula
encounter_type: subsequent encounter
severity: type IIIA||type IIIB||type IIIC
complication: malunion
laterality: left
Relations:
(Displaced oblique fracture||shaft)
(shaft||fibula)
(left||fibula)
(Displaced oblique fracture||subsequent encounter)
(subsequent encounter||open fracture)
(open fracture||type IIIA)
(open fracture||type IIIB)
(open fracture||type IIIC)
(open fracture||malunion)
### Input
ICD Code Description: Maternal care for hydrops fetalis, second trimester, fetus 2
### Output
Entities:
condition: hydrops fetalis
trimester: second trimester
fetus: fetus 2
Relations:
(hydrops fetalis||second trimester)
(hydrops fetalis||fetus 2)"""
我们使用simple_icd_10_cm库来获取所有ICD代码及其相应的描述:
import simple_icd_10_cm as cm
from tqdm import tqdm
codes = cm.get_all_codes()
icd_code_description = {}
for item in tqdm(codes):
if cm.is_leaf(item):
icd_code_description[item] = cm.get_description(item)
我们实例化OpenAI客户端,并开始使用GPT-4o mini提取ICD代码的实体和关系。
from openai import OpenAI
client = OpenAI(
api_key="",
)
def get_completion(prompt, input, model="gpt-4o-mini", temperature=0.0):
response = client.chat.completions.create(
model=model,
messages=[{"role": "system", "content": prompt},
{"role": "user", "content": input}],
temperature=temperature,
)
return response.choices[0].message.content
extracted_graphs = {}
for key, value in tqdm(icd_code_description.items()):
input_code_description = "ICD Code Description: " + value
output = get_completion(prompt_relation_extraction, input_code_description)
extracted_graphs[key] = output
实体链接
在提取了实体和关系之后,我们通过将它们链接到统一医学语言系统(UMLS)来对所有实体进行规范化。UMLS由美国国立医学图书馆开发,整合了各种健康和生物医学词汇及标准,如SNOMED-CT和MeSH。每个医学实体都与一个称为唯一概念标识符(CUI)的唯一标识符相关联。
一旦与UMLS链接,我们就使用大型语言模型(LLM)提取的实体的规范名称。为了进行链接,我们使用Scispacy的实体链接器,该链接器基于3-gram字符相似性将实体与UMLS概念进行匹配。由于我们已经提取了实体,因此我们绕过了Scispacy的默认实体提取管道,并直接将提取的实体传递给CandidateGenerator对象。这个过程提供了可能的UMLS概念ID,我们从中选择与输入相似度得分最高的概念ID。
candidate_generator = CandidateGenerator(name="umls")"umls")
entity_linker = EntityLinker(resolve_abbreviations=True, name="umls", candidate_generator=candidate_generator)
def get_max_similarity_concept_id(candidates):
"""
Determines the concept ID with the maximum similarity score from a list of candidate concepts.
Args:
candidates (list): A list of MentionCandidate objects, each containing a 'similarities' list and a 'concept_id'.
Returns:
any: The concept ID associated with the highest similarity score in the given candidates list, or None if the list is empty.
"""
max_similarity = float('-inf')
best_concept_id = None
for candidate in candidates:
candidate_max_similarity = max(candidate.similarities)
if candidate_max_similarity > max_similarity:
max_similarity = candidate_max_similarity
best_concept_id = candidate.concept_id
return best_concept_id
def normalize_entities(entities):
"""
Extracts the top linked concept for the first recognized entity in a given text using scispaCy.
Parameters:
- text (str): The input text to extract entities from.
Returns:
- str: The normalized entity if identified, or the original entity text.
"""
candidates = candidate_generator(entities, k = 5)
normalized_entity_map = {}
for entity, candidate in tqdm(zip(entities, candidates)):
most_likely_cui = get_max_similarity_concept_id(candidate)
cui_entity = entity_linker.kb.cui_to_entity[most_likely_cui]
normalized_entity_map[entity] = cui_entity.canonical_name
return normalized_entity_map
all_entities = []
for key, value in tqdm(extracted_graphs.items()):
entity_list = extract_entities(value)
all_entities += entity_list
all_entities = list(set(all_entities))
normalized_entity_map = normalize_entities(all_entities)
构建知识图谱(KG)
在从每个ICD描述中提取了实体和关系之后,我们开始构建知识图谱。首先,我们解析来自大型语言模型(LLM)预测的所有实体和关系。接下来,我们将这些实体映射到它们的UMLS规范名称,以进行标准化,并将它们作为节点添加到图中。我们根据LLM预测的关系,在共享关系的节点之间创建边。
此外,我们还在图中添加了一个代表ICD代码的节点,并在该节点与每个提取的实体之间创建边,这有助于未来在需要检索ICD代码时进行图查询。我们使用NetworkX库来构建这个图。
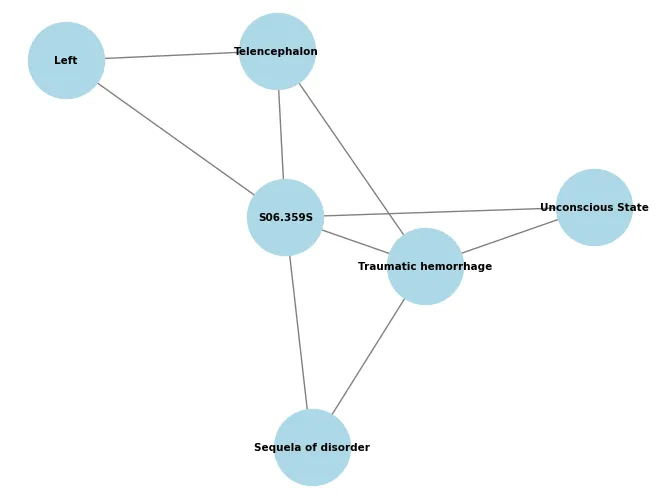
def parse_entities(lines, normalized_entity_map):
"""
Parses entity information from the provided lines.
Args:
lines (list): List of strings containing entity data in the format "EntityType: EntityName1 || EntityName2".
normalized_entity_map (dict): Dictionary mapping original entity names to their normalized forms.
Returns:
tuple: A tuple containing:
- entities (dict): A dictionary where keys are normalized entity names and values are entity types.
- overall_entities (set): A set of all entity names encountered.
"""
entities = {}
overall_entities = set()
for line in lines:
if ':' in line:
entity_type, entity_names = line.split(':', 1)
entity_type = entity_type.strip()
entity_names = [name.strip() for name in entity_names.split("||")]
for entity_name in entity_names:
overall_entities.add(entity_name)
entities[normalized_entity_map[entity_name]] = entity_type
return entities, overall_entities
def parse_relations(lines):
"""
Parses relation information from the provided lines.
Args:
lines (list): List of strings containing relation data in the format "(EntityName1 || EntityName2)".
Returns:
list: A list of tuples representing relations, where each tuple is a pair of related entity names.
"""
relations = []
for line in lines:
if line.startswith('(') and line.endswith(')'):
entity_names = [e.strip() for e in line[1:-1].split('||')]
if len(entity_names) == 2 and all(entity_names):
relations.append((entity_names[0], entity_names[1]))
return relations
def build_graph(input_text, icd_code, icd_description, normalized_entity_map):
"""
Builds a graph based on entity and relation information extracted from input text.
Args:
input_text (str): Text containing entity and relation information.
icd_code (str): ICD code for the graph root node.
icd_description (str): Description of the ICD code.
normalized_entity_map (dict): Dictionary mapping original entity names to their normalized forms.
Returns:
networkx.Graph: A NetworkX graph representing the entities, relations, and the ICD root node.
"""
lines = input_text.strip().split('\n')
entities_section, relations_section = split_sections(lines)
entities, overall_entities = parse_entities(entities_section, normalized_entity_map)
relations = parse_relations(relations_section)
G = create_graph(icd_code, icd_description, entities, overall_entities, relations, normalized_entity_map)
return G
def split_sections(lines):
"""
Splits the input lines into separate sections for entities and relations.
Args:
lines (list): List of strings containing the lines of the input text.
Returns:
tuple: A tuple containing:
- entities_section (list): List of strings corresponding to the "Entities:" section.
- relations_section (list): List of strings corresponding to the "Relations:" section.
"""
entities_section = []
relations_section = []
current_section = None
for line in lines:
line = line.strip()
if line == 'Entities:':
current_section = entities_section
elif line == 'Relations:':
current_section = relations_section
elif line and current_section is not None:
current_section.append(line)
return entities_section, relations_section
def create_graph(icd_code, description, entities, overall_entities, relations, normalized_entity_map):
"""
Creates a NetworkX graph using the provided ICD code, entities, and relations.
Args:
icd_code (str): ICD code for the root node of the graph.
description (str): Description of the ICD code.
entities (dict): Dictionary of entities with their types.
overall_entities (set): Set of all entity names encountered.
relations (list): List of tuples representing relations between entities.
normalized_entity_map (dict): Dictionary mapping original entity names to their normalized forms.
Returns:
networkx.Graph: A NetworkX graph representing the entities, relations, and the ICD root node.
"""
G = nx.Graph()
G.add_node(icd_code, type="ICD", description = description)
for entity_name, entity_type in entities.items():
G.add_node(entity_name, type=entity_type)
G.add_edge(entity_name, icd_code)
for entity1, entity2 in relations:
if entity1 in overall_entities and entity2 in overall_entities:
entity1_normalized = normalized_entity_map[entity1]
entity2_normalized = normalized_entity_map[entity2]
G.add_edge(entity1_normalized, entity2_normalized)
return G
graphs_list = []
for key, value in tqdm(extracted_graphs.items()):
icd_description = icd_code_description[key]
graph = build_graph(value, key, icd_description, normalized_entity_map)
graphs_list.append(graph)
kg = nx.compose_all(graphs_list)
在为每个代码构建了图之后,我们将所有图链接成一个更大的单一知识图谱(KG),该图谱现在表示所有的ICD代码。
GraphDB 索引
我们使用Neo4j (一种流行的图数据库)来为我们的图建立索引。对于我们的用例,我们利用Neo4j Aura在线提供的免费实例。我们首先连接到我们的实例,然后使用Python中的neo4j包以批处理的方式为我们的节点和关系建立索引。
from neo4j import GraphDatabase
import networkx as nx
BATCH_SIZE = 1000
uri = ""
username = ""
password = ""
def create_nodes(tx, nodes):
"""
Creates or updates nodes in the Neo4j database.
This function takes a list of nodes, and for each node, it either creates a new node or updates an existing one based on the node's `id`. The attributes of each node are set or updated as specified in the `attributes` field.
Args:
tx (neo4j.Transaction): The active Neo4j transaction.
nodes (list): A list of dictionaries, where each dictionary represents a node with the following structure:
- 'id': The unique identifier for the node.
- 'attributes': A dictionary of key-value pairs representing node attributes.
"""
tx.run(
"""
UNWIND $nodes AS node
MERGE (n:Node {id: node.id})
SET n += node.attributes
""",
nodes=nodes
)
def create_relationships(tx, relationships):
"""
Creates or updates relationships between nodes in the Neo4j database.
This function takes a list of relationships and for each relationship, it either creates a new relationship or updates an existing one
based on the source and target node identifiers (`source_id` and `target_id`). The relationship's attributes are set or updated
as specified in the `attributes` field.
Args:
tx (neo4j.Transaction): The active Neo4j transaction.
relationships (list): A list of dictionaries, where each dictionary represents a relationship with the following structure:
- 'source_id': The unique identifier of the source node.
- 'target_id': The unique identifier of the target node.
- 'attributes': A dictionary of key-value pairs representing relationship attributes.
"""
tx.run(
"""
UNWIND $relationships AS rel
MATCH (a:Node {id: rel.source_id})
MATCH (b:Node {id: rel.target_id})
MERGE (a)-[r:RELATES_TO]-(b)
SET r += rel.attributes
""",
relationships=relationships
)
def create_index(tx):
"""
Creates a unique constraint on the `id` property of the `Node` label in the Neo4j database.
This function ensures that the `id` property of each `Node` is unique, preventing the creation of nodes
with duplicate `id` values. It is typically used for enforcing data integrity and speeding up lookup operations.
Args:
tx (neo4j.Transaction): The active Neo4j transaction.
"""
tx.run("CREATE CONSTRAINT FOR (n:Node) REQUIRE n.id IS UNIQUE")
driver = GraphDatabase.driver(uri, auth=(username, password))
with driver.session() as session:
session.write_transaction(create_index)
nodes = [
{'id': node_id, 'attributes': attributes}
for node_id, attributes in kg.nodes(data=True)
]
relationships = [
{
'source_id': source_id,
'target_id': target_id,
'attributes': attributes
}
for source_id, target_id, attributes in kg.edges(data=True)
]
for i in range(0, len(nodes), BATCH_SIZE):
batch = nodes[i:i+BATCH_SIZE]
session.write_transaction(create_nodes, batch)
for i in range(0, len(relationships), BATCH_SIZE):
batch = relationships[i:i+BATCH_SIZE]
session.write_transaction(create_relationships, batch)
driver.close()
查询图形
在将我们的图形索引到Neo4j之后,我们就可以开始使用Cypher (Neo4j的用于图形数据交互的声明式查询语言)来查询它了。Cypher为我们提供了一种强大且灵活的图形查询方法。
在我们的初步示例中,我们查询了所有与可能发生在右眼睑上的撕裂伤相关的ICD代码。在这里,“撕裂伤”代表状况,“眼睑”代表身体部位,“右侧”表示侧别。关系将状况与身体部位以及侧别与身体部位连接起来。我们将这些约束条件编码到Cypher查询中,从而能够检索到相关的结果。
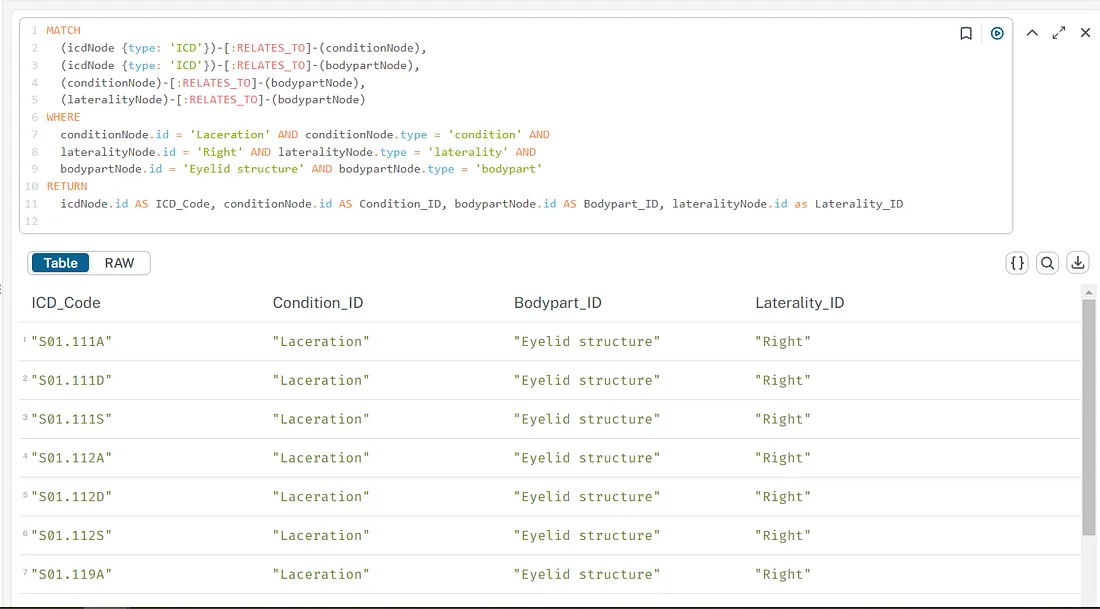
查询成功检索到了符合指定条件的相关ICD代码。我们还尝试了更灵活的Cypher查询,特别是通过检索与“撕裂伤”状况相关的所有ICD代码。通过以这种方式放宽约束条件,我们能够捕获到更广泛的相关ICD代码集,从而为此状况找到了多个相关匹配项。
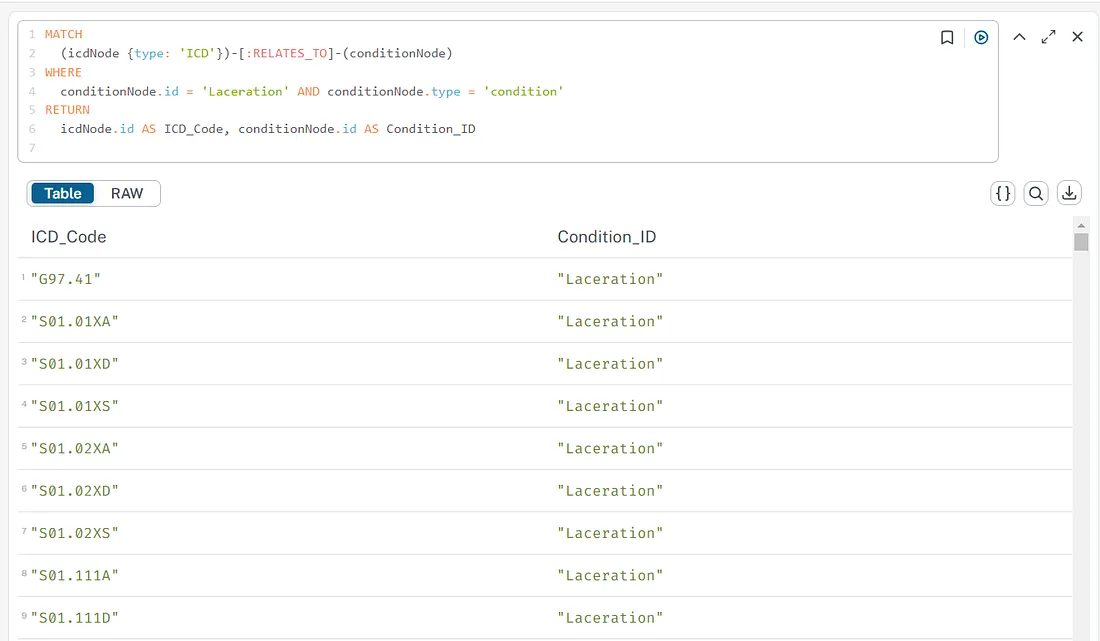
通过为实体之间的关系添加特定约束,我们可以创建更有针对性和精确的查询。此外,Cypher还提供了灵活的功能,如自由文本搜索(例如,使用CONTAINS关键字)、不区分大小写的匹配以及使用可选匹配来处理不同场景。
结论
在这篇文章中,我们探索了如何将ICD代码表示为结构化的知识图谱(KG),以及如何对其进行索引和查询。以这种方式使用知识图谱可以让我们在检索增强生成(RAG)设置中与大型语言模型(LLM)进行更深入的集成,从而帮助我们生成更基于事实的输出。