改进RAG准确率:微调领域知识嵌入模型(第2部分)
这是本系列的第 2 部分,在第 1 部分中,我们研究了如何在句子转换器库的帮助下使用预训练模型微调嵌入模型。在第 2 部分中,我们将研究这种微调在构建 RAG 管道时如何发挥作用,特别是在检索步骤和最终生成阶段。
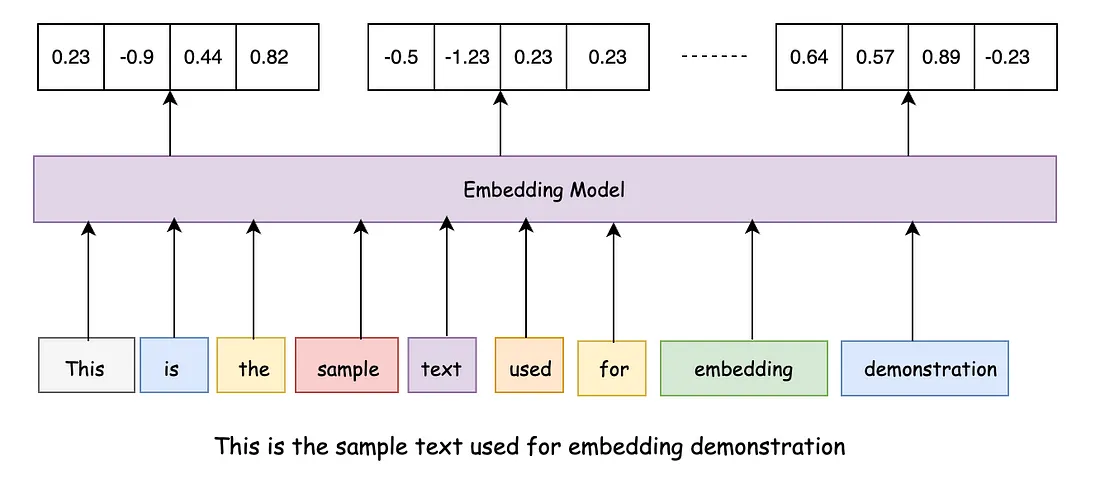
简介
本系列第二部分介绍的架构旨在通过利用两个不同的模型(一个微调嵌入模型和一个预训练嵌入模型)高效地将领域特定数据摄入向量存储中。这两个模型协同工作,处理输入数据,确保有效捕获和存储相关上下文信息以供检索。此设置的主要目标是提高语言模型在用户与系统交互时生成响应的准确性和上下文理解能力。
为实现这一目标,每个用户查询都会通过微调模型和预训练模型进行处理,这两个模型从查询中提取有意义的表示,并检索最相关的上下文信息。然后,将检索到的上下文传递给一个大型语言模型(LLM),该模型具有两个主要功能。首先,它根据检索到的知识生成响应,确保答案与领域特定语料库保持一致。其次,LLM充当评估器,使用两个关键指标(“答案相关性得分”和“上下文相关性得分”)来评估生成响应的质量。这些评估提供了有关检索到的上下文与查询的匹配程度以及生成响应的相关性和准确性的见解。
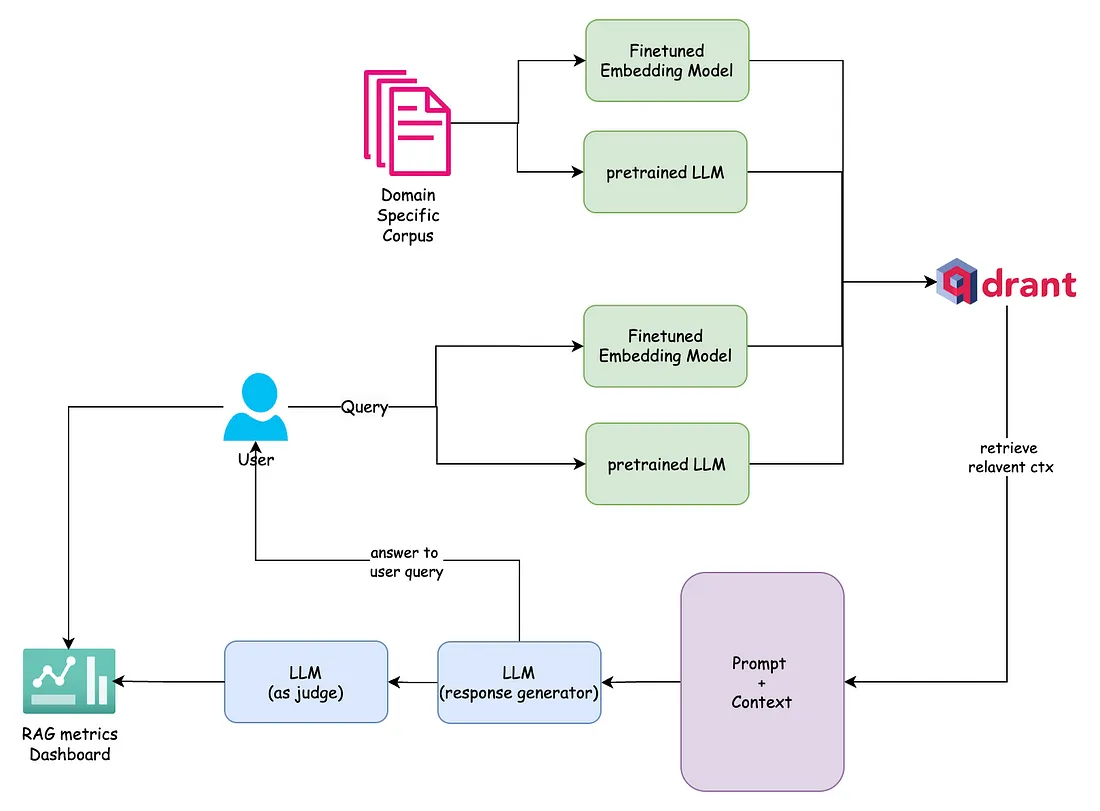
评估分数随后被编译并展示在一个结构化的仪表板上,以便持续监控和优化系统的性能。通过以可视化格式呈现这些分数,决策者可以分析趋势、识别潜在的改进领域,并据此微调检索和响应生成机制。这种架构不仅提高了响应的质量,还提供了一种结构化的方法来评估和优化呈现给用户的信息的相关性。
通过这种迭代方法,系统确保领域特定的查询能够以更高的精度得到处理,使其特别适用于需要深入理解上下文的应用程序,如企业搜索解决方案、技术文档检索和知识管理系统。精调的嵌入表示、预训练模型和基于大型语言模型(LLM)的评估相结合,构建了一个强大的管道,增强了检索和响应生成过程,最终打造出一个更智能、更懂上下文的问题回答系统。
实现
让我们开始代码实现。以下是项目结构。
.
├── biomedical_rag.py
├── data
│ └── pubmed_knowledge.pdf
├── logger_util.py
├── logs
│ └── app.log
├── pre_processing.py
└── requirements.txt
让我们从日志工具开始,该工具将把代码中流动的所有事件记录到日志文件中,同时也会在控制台输出。
import logging
import os
import datetime
import sys
class LoggerUtil:
"""A simple logging utility for Python applications."""
def __init__(self, name="app", log_level=logging.INFO, log_file=None, console_output=True):
"""
Initialize the logger utility.
Args:
name (str): Name of the logger
log_level (int): Logging level (e.g., logging.DEBUG, logging.INFO)
log_file (str, optional): Path to log file. If None, no file logging.
console_output (bool): Whether to output logs to console
"""
self.logger = logging.getLogger(name)
self.logger.setLevel(log_level)
self.logger.handlers = [] # Clear any existing handlers
# Create formatter
formatter = logging.Formatter(
'%(asctime)s - %(name)s - %(levelname)s - %(message)s',
datefmt='%Y-%m-%d %H:%M:%S'
)
# Add console handler if requested
if console_output:
console_handler = logging.StreamHandler(sys.stdout)
console_handler.setFormatter(formatter)
self.logger.addHandler(console_handler)
# Add file handler if log_file is provided
if log_file:
os.makedirs(os.path.dirname(log_file), exist_ok=True)
file_handler = logging.FileHandler(log_file)
file_handler.setFormatter(formatter)
self.logger.addHandler(file_handler)
def debug(self, message):
"""Log a debug message."""
self.logger.debug(message)
def info(self, message):
"""Log an info message."""
self.logger.info(message)
def warning(self, message):
"""Log a warning message."""
self.logger.warning(message)
def error(self, message):
"""Log an error message."""
self.logger.error(message)
def critical(self, message):
"""Log a critical message."""
self.logger.critical(message)
上面的 LoggerUtil 类是一个简单且灵活的 Python 应用程序日志工具。它使用给定的名称、日志级别以及可选的文件日志记录来配置日志记录器,同时确保消息带有时间戳格式。它支持同时向控制台和指定的日志文件记录日志。该类提供了用于记录不同严重性级别消息的方法,例如调试、信息、警告、错误和严重错误。此工具简化了日志设置,并确保了高效的消息跟踪,以便于调试和监控。
注:为了创建基于 PubMed 的知识 PDF,我们将使用 Hugging Face 上的“qiaojin/PubMedQA”数据集。
from datasets import load_dataset
from reportlab.lib.pagesizes import letter
from reportlab.pdfgen import canvas
from logger_util import LoggerUtil
import logging
# Create a logger that logs to both console and file
log = LoggerUtil(
name="pre_processing",
log_level=logging.DEBUG,
log_file="logs/app.log"
)
log.info("downloading and making data frame")
ds = load_dataset("qiaojin/PubMedQA", "pqa_artificial")
df = ds["train"].to_pandas()
def extract_contexts(num_of_records: int = 5000):
log.info("extract_contexts started")
# Limit to the first num_records
df_subset = df.head(num_of_records)
extracted_contexts = []
for i, row in df_subset.iterrows():
try:
# Based on the screenshot, it appears 'context' is already a dictionary
# with a 'contexts' key that contains a list of strings
contexts = row['context']['contexts'].tolist()
if isinstance(contexts, list):
context_text = ' '.join(contexts)
extracted_contexts.append(context_text)
else:
log.info(f"Warning: 'contexts' field is not a list in row {i}")
except (KeyError, TypeError) as e:
log.error(f"Error processing row {i}: {e}")
return extracted_contexts
def _wrap_text(text, canvas, max_width):
log.info(f"text styling for pdf started")
"""Break text into lines that fit within max_width."""
words = text.split()
lines = []
current_line = []
for word in words:
test_line = ' '.join(current_line + [word])
width = canvas.stringWidth(test_line)
if width <= max_width:
current_line.append(word)
else:
# If the line has content, add it
if current_line:
lines.append(' '.join(current_line))
current_line = [word]
# If a single word is too long, force it onto its own line
else:
lines.append(word)
# Don't forget the last line
if current_line:
lines.append(' '.join(current_line))
return lines
def create_pdf_doc(data, output_filename="data/pubmed_knowledge.pdf"):
log.info(f"creation of pdf started")
# Create a canvas with letter size
c = canvas.Canvas(output_filename, pagesize=letter)
width, height = letter
# Set up text area dimensions
margin = 50
text_width = width - 2 * margin
# Starting position and line height
x = margin
y = height - margin
line_height = 14
for string in data:
# Create a text object for wrapping text
textobject = c.beginText()
textobject.setTextOrigin(x, y)
# Set the wrap width
textobject.setWordSpace(0.1)
# Add the wrapped text
for line in _wrap_text(string, c, text_width):
textobject.textLine(line)
# Reduce available height
y -= line_height
# Check if need new page
if y < margin:
c.drawText(textobject)
c.showPage()
y = height - margin
textobject = c.beginText()
textobject.setTextOrigin(x, y)
# Draw the text object
c.drawText(textobject)
y -= line_height # Extra space between paragraphs
# Save the PDF
c.save()
log.info(f"PDF created successfully: {output_filename}")
# print("initializing extraction")
# data = extract_contexts()
# print("started pdf creation")
# create_pdf_doc(data=data)
这个“create_pdf_doc”、“_wrap_text”和“extract_contexts”脚本用于处理并从“qiaojin/PubMedQA”数据集中提取文本数据,然后生成格式化的PDF文档。它使用LoggerUtil工具记录消息,以便跟踪执行进度和潜在问题。数据集被加载到Pandas DataFrame中,extract_contexts函数从前5000条记录中检索并连接“contexts”字段,同时优雅地处理错误。
为了生成PDF,_wrap_text确保文本行通过拆分成适当大小的段落来适应页面宽度。然后,create_pdf_doc函数接收提取的文本,将其组织成可读的格式,并使用ReportLab将其写入PDF文件。它保持页边距,处理分页,并确保文本结构化的流动。在整个过程中,日志消息提供了每个步骤的见解,使调试和监控更加容易。
import os
from llama_index.core import VectorStoreIndex, SimpleDirectoryReader, Settings, StorageContext
from llama_index.core.query_engine import FLAREInstructQueryEngine
from llama_index.llms.anthropic import Anthropic
from llama_index.embeddings.huggingface import HuggingFaceEmbedding
from llama_index.vector_stores.qdrant import QdrantVectorStore
from qdrant_client import QdrantClient, AsyncQdrantClient
from qdrant_client.http.models import VectorParams, Distance
from dotenv import load_dotenv, find_dotenv
from deepeval.integrations.llama_index import (
DeepEvalAnswerRelevancyEvaluator,
DeepEvalContextualRelevancyEvaluator
)
from pre_processing import df
import nest_asyncio
from logger_util import LoggerUtil
import logging
# Create a logger that logs to both console and file
log = LoggerUtil(
name="biomedical_rag",
log_level=logging.DEBUG,
log_file="logs/app.log"
)
# Initialize nest_asyncio
nest_asyncio.apply()
load_dotenv(find_dotenv())
log.info("started with global settings")
COLLECTION_NAME = os.environ.get("QDRANT_COLLECTION_NAME")
Settings.chunk_size = int(os.environ.get("CHUNK_SIZE"))
Settings.chunk_overlap = int(os.environ.get("CHUNK_OVERLAP"))
Settings.embed_model = HuggingFaceEmbedding(model_name=os.environ.get("EMBEDDING_MODEL_ID"), device="mps",
trust_remote_code=True)
Settings.llm = Anthropic(model=os.environ.get("ANTHROPIC_MODEL_ID"), api_key=os.environ.get("ANTHROPIC_API_KEY"))
q_client = QdrantClient(url=os.environ.get("QDRANT_URL"), api_key=os.environ.get("QDRANT_API_KEY"))
async_q_client = AsyncQdrantClient(url=os.environ.get("QDRANT_URL"), api_key=os.environ.get("QDRANT_API_KEY"))
vector_store = QdrantVectorStore(collection_name=COLLECTION_NAME, client=q_client, aclient=async_q_client,
dense_config=VectorParams(size=int(os.environ.get("EMBEDDING_DIM")), distance=Distance.COSINE))
storage_ctx = StorageContext.from_defaults(vector_store=vector_store)
vector_store_index: VectorStoreIndex
docstore = None
if q_client.collection_exists(collection_name=COLLECTION_NAME):
log.info("creating vector index from existing collection")
vector_store_index = VectorStoreIndex.from_vector_store(vector_store=vector_store)
else:
log.info("collection doesn't exist hence creating and then indexing the data.")
docs = SimpleDirectoryReader(input_dir="data", required_exts=[".pdf"]).load_data(show_progress=True)
vector_store_index = VectorStoreIndex.from_documents(
documents=docs,
# our dense embedding model
embed_model=Settings.embed_model,
storage_context=storage_ctx,
)
evals_questions = []
def compute_question_answer_evals():
for i, row in df[:10].iterrows():
ctxs = []
question = row["question"]
# answer = row["long_answer"]
# evals_questions.append((question, answer))
log.info(f"questions {i} for evals: {question}")
query_engine = vector_store_index.as_query_engine(similarity_top_k=50, response_mode='tree_summarize')
# flare_query_engine = FLAREInstructQueryEngine(
# query_engine=query_engine,
# max_iterations=5,
# verbose=True
# )
response = query_engine.query(str_or_query_bundle=question)
for node in response.source_nodes:
ctxs.append(node.text)
ans_rel_evaluator = DeepEvalAnswerRelevancyEvaluator(threshold=0.7, model=os.environ.get("OPENAI_MODEL_ID"))
ctx_rel_evaluator = DeepEvalContextualRelevancyEvaluator(threshold=0.7, model=os.environ.get("OPENAI_MODEL_ID"))
ans_rel_result = ans_rel_evaluator.evaluate(response=response.response, query=question, contexts=ctxs)
ctx_rel_result = ctx_rel_evaluator.evaluate(response=response.response, query=question, contexts=ctxs)
log.info(f"Response: {response}")
log.info(f"Answer relevancy: {ans_rel_result}")
log.info(f"Context relevancy: {ctx_rel_result}")
log.info("="*200)
compute_question_answer_evals()
该脚本使用llama_index、Qdrant、Anthropic和gpt-4o实现了一个生物医学检索增强生成(RAG)系统,用于问答和评估。它首先通过LoggerUtil配置日志记录以跟踪执行过程。加载环境变量以设置参数,如块大小、嵌入模型和Qdrant集合名称。脚本初始化nest_asyncio以允许嵌套事件循环,从而能够异步执行操作。
系统的核心涉及使用Qdrant创建或加载向量存储。如果指定的集合已存在,则从中构建VectorStoreIndex;否则,脚本从“data”目录读取PDF文档,使用Hugging Face模型对其进行嵌入,并将其索引到Qdrant中。这个向量索引允许高效地检索问答所需的相关信息。
compute_question_answer_evals函数处理数据集的前十条记录,提取问题,并使用基于相似度的检索方法查询向量存储索引。然后,使用DeepEvalAnswerRelevancyEvaluator和DeepEvalContextualRelevancyEvaluator对检索到的上下文进行评估,评估答案和检索到的上下文与给定问题的相关性。结果(包括检索到的响应、答案相关性得分和上下文相关性得分)被记录以供进一步分析。此设置提供了一个自动化管道,用于评估生物医学知识检索和响应生成的有效性。
指标可视化:
最后,将前一过程的日志发送到大型语言模型(LLM),以获取可视化所需的数据和数据的关键亮点。将数据发送到LLM并根据我们的需求和关键亮点获取数据的这一过程将留待第三部分进行。现在,我们将看到指标的可视化方式以及仪表板的外观。

实验与指标研究:
实验在三个方面存在差异:
1. 块大小:使用了两个值,分别为128和512。
2. 块重叠:使用了两个值,分别为50和100。
3. 嵌入模型:测试了三种不同的嵌入模型:
- pavanamantha/distilroberta-pubmed-embeddings
- sentence-transformers/all-MiniLM-L6-v2
- pavanamantha/bge-base-en-biomed
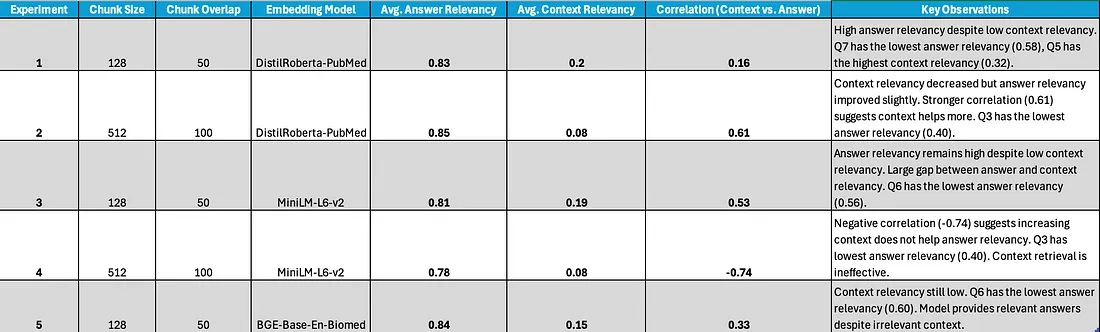
实验的关键发现
块大小的影响(128 vs. 512)
- 将块大小从128增加到512(实验1→2和3→4)导致:
- 上下文相关性降低(实验1&2中从0.20降至0.08,实验3&4中从0.19降至0.08)。
- 实验2中相关性较高(0.61),但实验4中呈负相关(-0.74),表明分块的影响因嵌入模型而异。
- 实验2中答案相关性略有提高,但实验4中有所下降。
块重叠的影响(50 vs. 100)
- 较高的重叠(实验2和4)并不一定会改善检索效果。
- 在实验4中,负相关(-0.74)表明,在某些情况下,更多的重叠可能会损害检索效果。
嵌入模型的影响
DistilRoberta-PubMed(实验1&2)
- 产生较高的答案相关性(~0.83–0.85),但上下文相关性较低。
- 块大小增加时相关性较高(0.61)。
MiniLM-L6-v2(实验3&4)
- 显示出较高的答案相关性(~0.78–0.81),但在较大块大小时表现不佳(实验4,相关性为-0.74)。
BGE-Base-En-Biomed(实验5)
- 在两者之间取得平衡,保持良好的答案相关性(0.84)。
- 相关性(0.33)表明,与其他模型相比,上下文稍微更有用。
结论
我们的实验表明,尽管上下文相关性较低,但微调后的嵌入(DistilRoberta-PubMed和BGE-Base-En-Biomed)始终能产生较高的答案相关性(~0.83–0.85)。其中,使用较大分块的DistilRoberta-PubMed(实验2)取得了最高的相关性(0.61),表明增加块大小有时可以提高检索效果。
相比之下,预训练模型(MiniLM-L6-v2)在分块方面表现不佳,实验4显示出负相关(-0.74),表明添加更多上下文实际上降低了答案质量。
本系列上文:改进RAG准确率:微调领域知识嵌入模型(第1部分)