使用python中的Numpy进行t检验
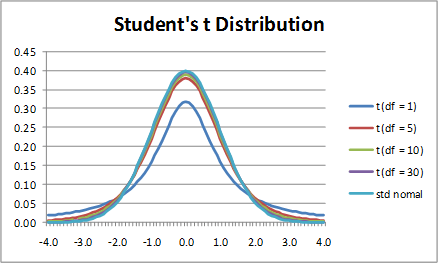
虽然像SciPy和PyMC3这样的流行的统计数据库有预定义的函数来计算不同的测试,但是为了了解这个过程的数学原理,必须了解后台的运行。本系列将帮助你了解不同的统计测试,以及如何在python中只使用Numpy执行它们。
t检验是统计学中最常用的程序之一。但是,即使是经常使用t检验的人,也往往不清楚当他们的数据转移到后台使用像Python和R的来操作时会发生什么。
什么是t检验
t检验(Student's T Test)比较两个平均值(均值),然后告诉你它们彼此是否有差异。并且,t检验还会告诉你这个差异有没有意义,换句话说,它让你知道这些差异是否可能是偶然发生的。
举一个非常简单的例子:假设你得了感冒,你尝试了自然疗法。你的感冒持续了几天。下一次感冒时,你买了一个非处方药,感冒了一周。你调查你的朋友,他们都告诉你,当他们采取同种方法治疗时,他们的感冒时间较短(平均 3天)。你真正想知道的是,这种情况会不会是碰巧的?t测试可以通过比较两组的方法来回答你,让你知道这些结果碰巧发生的概率。
再举一个例子:t检验可以用在现实生活中作为比较手段。例如,一家制药公司可能想要测试一种新的抗癌药,以确定它是否能提高预期寿命。在实验中,会有一个对照组(给予安慰剂或“糖丸”的组)。对照组可能显示平均寿命增长5年,而服用新药平均寿命增长6年。看样子药物可能产生了效果。但这也可能是个巧合。为了验证这一点,研究人员将使用t检验来确定整这样的情况会不会一直发生。
什么是t分数
t分数是两个组之间的差值与组内差的比值。t分数越大,组间的差异越大。t分数越小,组间的相似度就越大。t分数为3代表这些组是彼此之间的三倍。当你运行t-score时,t值越大,结果越可能重复。
- t分数越大,这些组差异越大。
- 如果t分数越小,这些组越相似的。
什么是T值和P值
“足够大”多大?每个t值都有伴随着一个p值。p值是你的样本数据的结果偶然发生的概率。P值为0%至100%。它们通常写为小数。例如,5%的p值为0.05。低p值好;低假定值是好的;他们指出你的数据不是偶然发生的。例如,p值为0.1意味着实验结果只有1%的可能是碰巧发生的。多数情况下,p值为0.05(5%)表示数据有效。
t检验有哪些类型
t检验有三种主要类型:
1.独立样本t检验:比较两组平均值的方法。
2.配对样本t检验:比较同一组中不同时间(例如,相隔一年)平均值的方法。
3.单一样本t检验:检验单个组的平均值对照一个已知的平均值。
如何执行2个样本的t检验
假设,我们必须检验人口中男性的身高与女性的身高是否不同。我们从人口中抽取样本,并使用t检验来判断结果是否有效。
步骤:
1.确定一个虚无假设和对立假设
一般来说,零假设将表明被测试的两个群体没有统计学意义上的差异。对立假设将说明有差异存在。在这个例子中我们可以说:
虚无假设:男女平均身高相同
对立假设:男女平均身高不相同
2.收集样本数据
下一步是为每个群体收集一组数据。在我们的示例中,我们收集了2组数据即:女性身高和男性身高。理想情况下样本量应该是相同的,但这显然不现实。让我们设定样本大小分别是nx和ny。
3.确定置信区间和自由度
这就是我们所说的alpha(α)。α的代表值为0.05。这意味着这个测试的结论有效的可能性是95%。自由度可以通过以下公式计算:
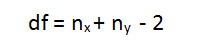
4.计算t统计
t统计量可以用下面的公式计算:
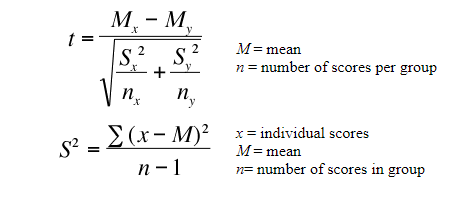
其中, Mx和My是男性和女性两个样本的平均值。
Nx和Ny是两个样本的样本空间
S是标准偏差
5.从t分布
计算临界t值为了计算临界t值,我们需要2件事,选择的α值和自由度。临界t值的公式是复杂的,但是固定的一对自由度和α的值是固定的。因此,我们使用一个表来计算临界t值:
在python中,我们将使用sciPy包中的函数计算而不是在表中查找。(我保证,这是我们唯一一次需要用它!)
6.将临界t值与计算出的t统计量进行比较
如果计算的t统计量大于临界t值,则该测试得出结论:两个群体之间存在统计上显著的差异。因此,你可以驳回虚无假设的两个人群之间没有统计学上显著差异结论。
在任何其他情况下,两个人群之间没有统计学上的显著差异。测试无法驳回虚无假设,但我们接受了对立假设,也就是说男性和女性的身高在统计学上是不同的。
代码如下:
## Import the packages
import numpy as np
from scipy import stats
## Define 2 random distributions
#Sample Size
N = 10
#Gaussian distributed data with mean = 2 and var = 1
a = np.random.randn(N) + 2
#Gaussian distributed data with with mean = 0 and var = 1
b = np.random.randn(N)
## Calculate the Standard Deviation
#Calculate the variance to get the standard deviation
#For unbiased max likelihood estimate we have to divide the var by N-1, and therefore the parameter ddof = 1
var_a = a.var(ddof=1)
var_b = b.var(ddof=1)
#std deviation
s = np.sqrt((var_a + var_b)/2)
s
## Calculate the t-statistics
t = (a.mean() - b.mean())/(s*np.sqrt(2/N))
## Compare with the critical t-value
#Degrees of freedom
df = 2*N - 2
#p-value after comparison with the t
p = 1 - stats.t.cdf(t,df=df)
print("t = " + str(t))
print("p = " + str(2*p))
#Note that we multiply the p value by 2 because its a twp tail t-test
### You can see that after comparing the t statistic with the critical t value (computed internally) we get a good p value of 0.0005 and thus we reject the null hypothesis and thus it proves that the mean of the two distributions are different and statistically significant.
## Cross Checking with the internal scipy function
t2, p2 = stats.ttest_ind(a,b)
print("t = " + str(t2))
print("p = " + str(2*p2))